RFdiffusion3 开源:更快、更强、更“万能”的通用分子生成模型
今天,千呼万唤始出来,华盛顿大学蛋白设计研究所(IPD)正式宣布:
RFdiffusion3(RFD3)正式开源!(最爱的游戏终于更新了)
RFdiffusion3是一个能够从原子级别生成全新蛋白结构的AI模型,它能让蛋白与DNA、小分子、酶底物等各种生物分子精准相互作用。
“我们构建它时的目标,是让科学界能用它去创造那些我们尚未想象的东西。”
—— Rohith Krishna, PhD,项目负责人
论文链接:https://doi.org/10.1101/2025.09.18.676967
开源代码链接:https://github.com/RosettaCommons/foundry
一切都在变:AI开始理解“化学细节”
几十年来,蛋白设计面临一个核心难题:
要让蛋白与特定DNA或化学底物结合,必须精确控制每个原子的空间位置。
这对于酶设计尤其困难,因为催化中心中的原子必须排列到“化学键”级精度。

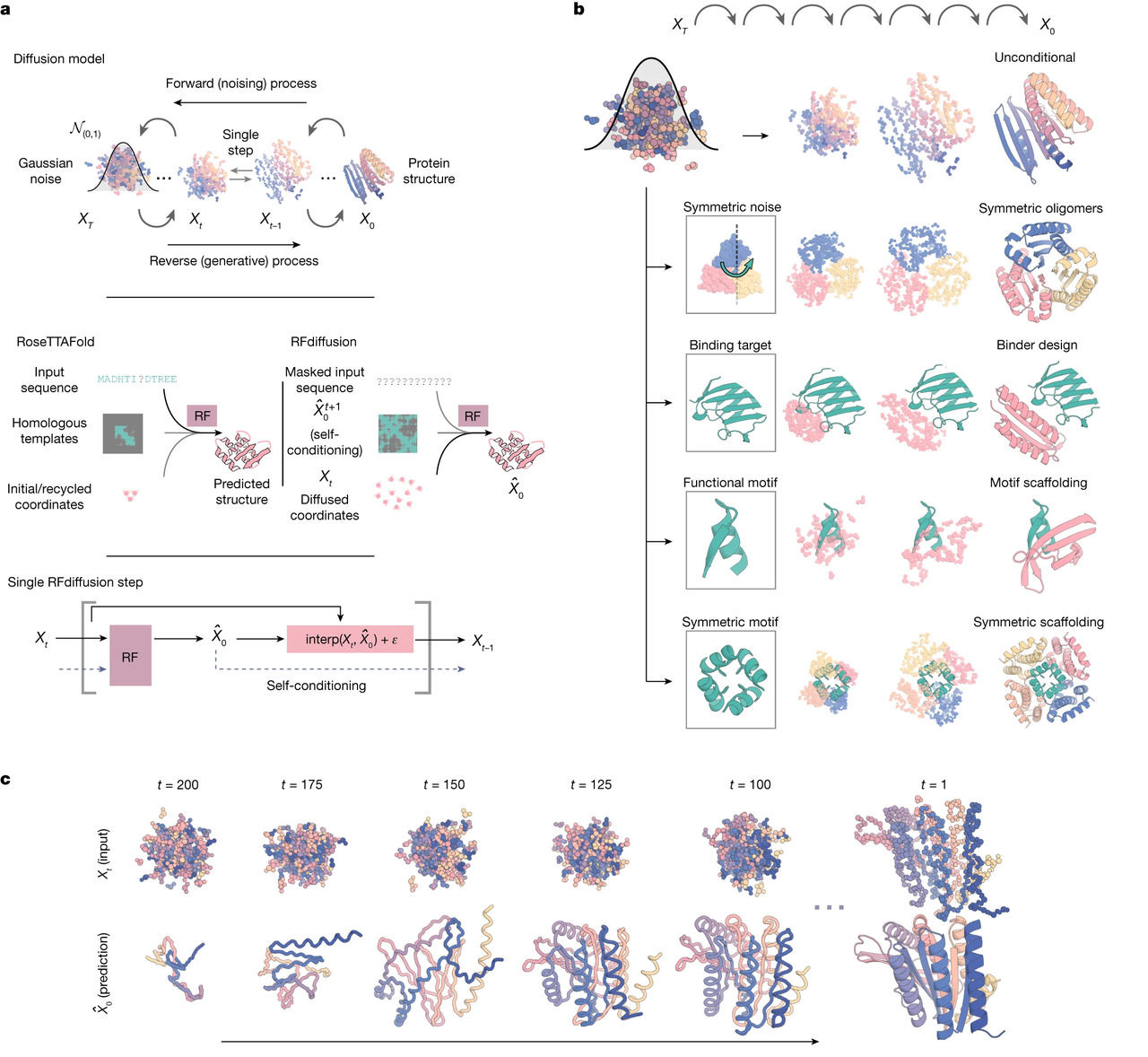
RFdiffusion 1
过去的AI工具(包括RFdiffusion1和2)试图通过简化建模来降低难度,但这种简化也让它们难以真正“理解”分子的化学相互作用。RFdiffusion2也在昨天publish在Nature methods上,感兴趣的读者可以去看看原文(https://www.nature.com/articles/s41592-025-02975-x),或者看我之前写的解读文章:

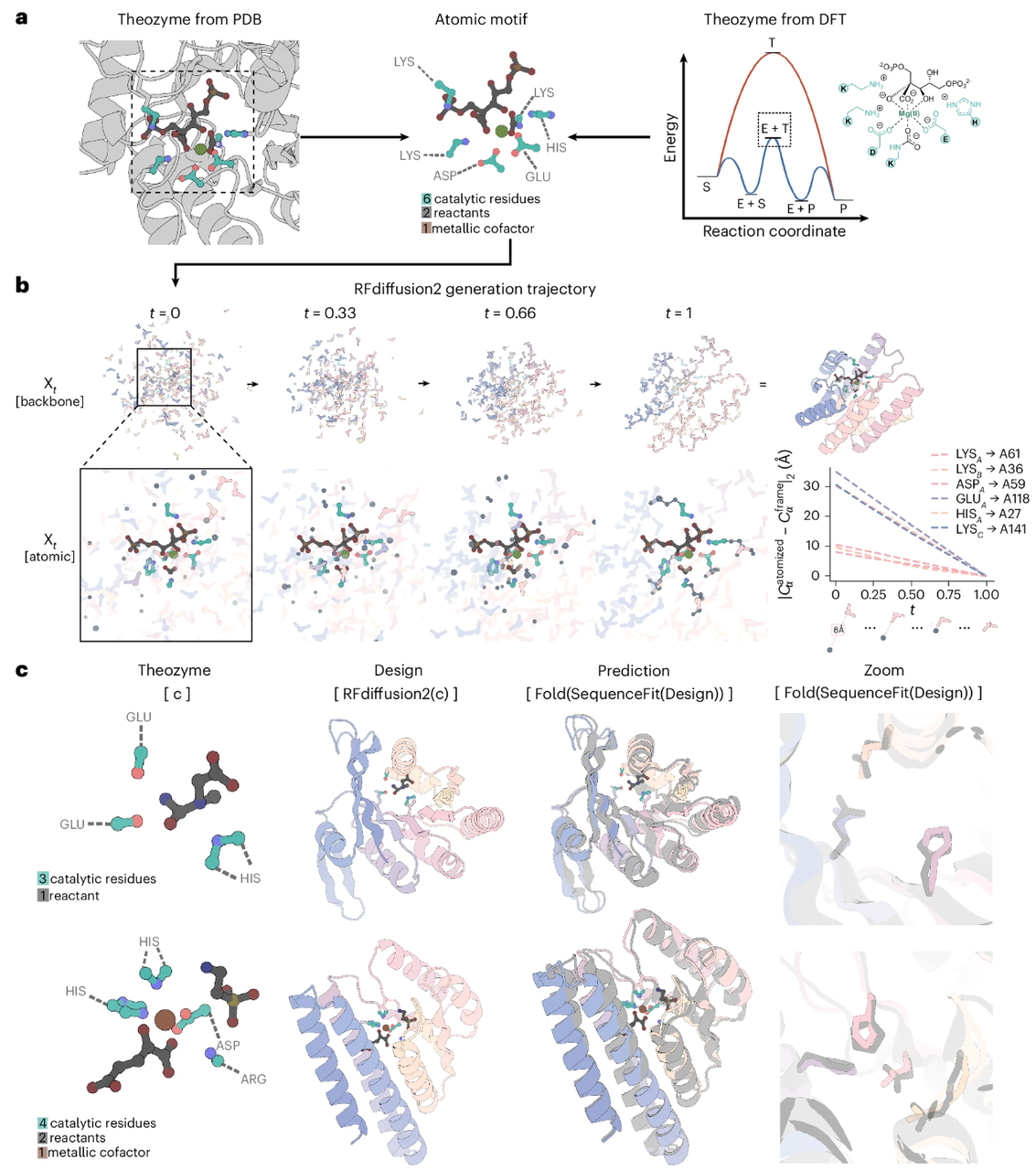
RFdiffusion2
RFdiffusion3 则完全不同。
它直接以“原子为基本单位”进行扩散生成,能够以前所未有的精度捕捉化学细节。
“核心创新是精确控制。
我们终于能告诉模型:哪些化学作用最重要,然后它会自己构建出能实现这些作用的蛋白。”
—— Jasper Butcher, PhD,模型首席开发者
关于RFdiffusion3的详细介绍,可以看我之前写的推送:

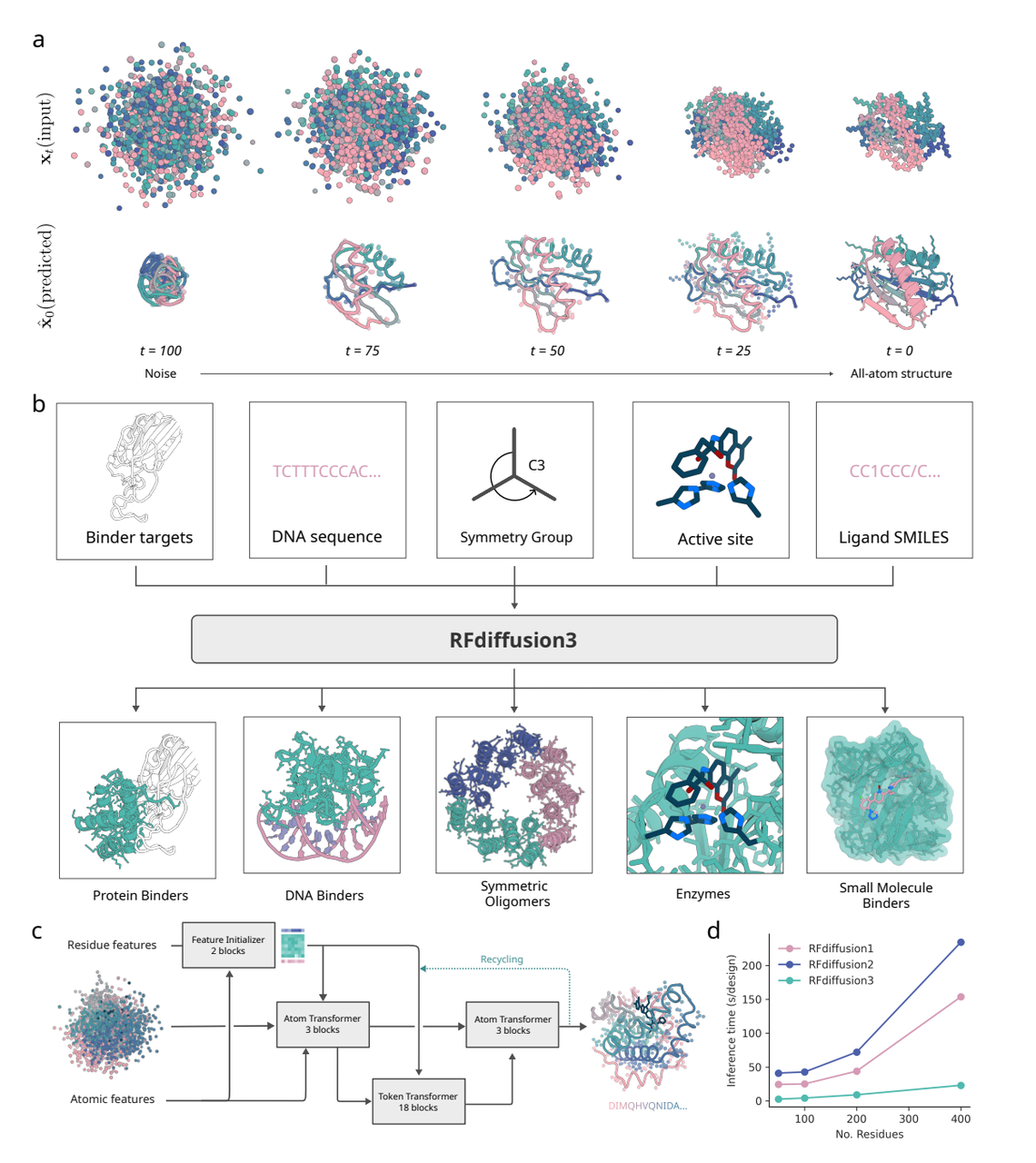
RFD3为什么值得关注——三大关键突破
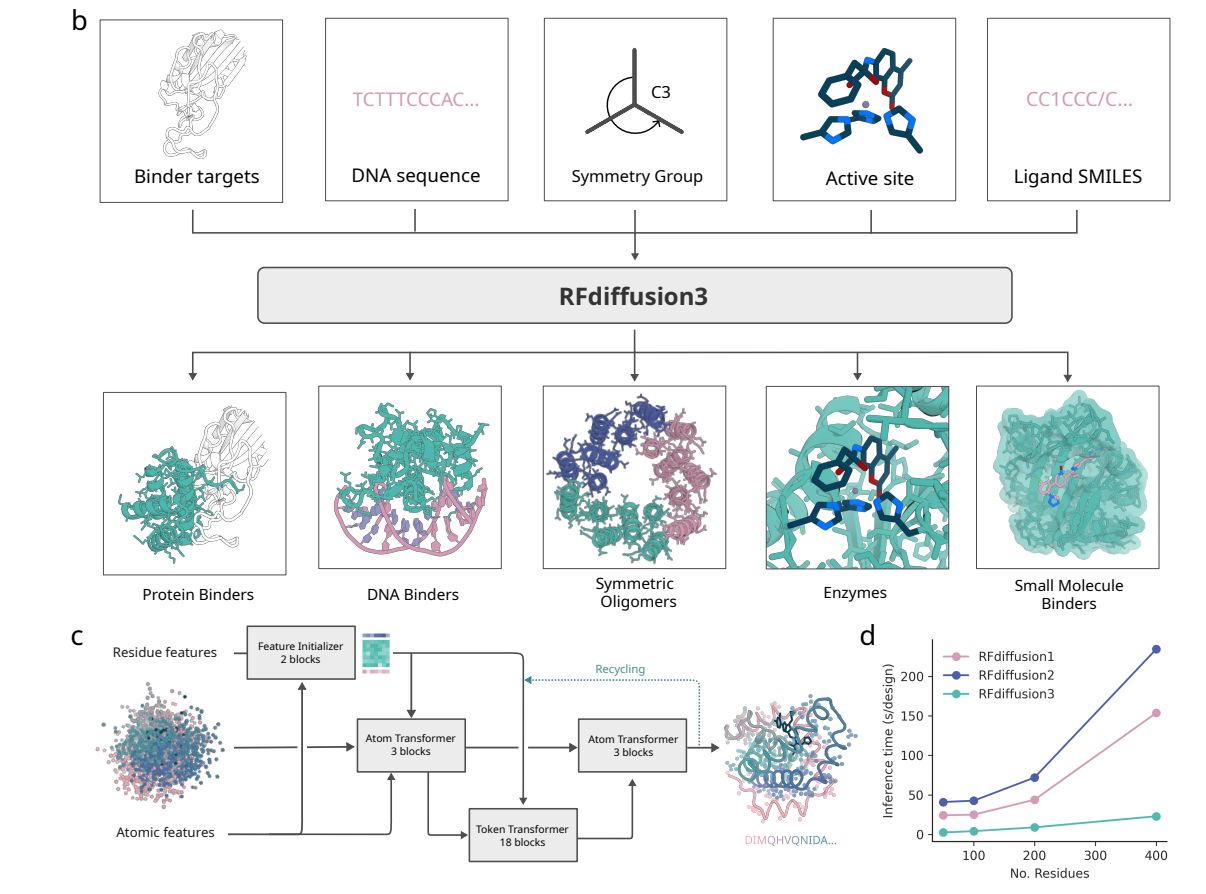
1️⃣ 原子级扩散建模(Atom-level Diffusion)
RFD3不再以残基为最小单元,而是直接在每个原子坐标上进行生成。
它能自动生成氢键网络、溶剂可及性分布、酶活性位点、质心约束、对称结构,让设计者能够像编程一样控制分子几何 。
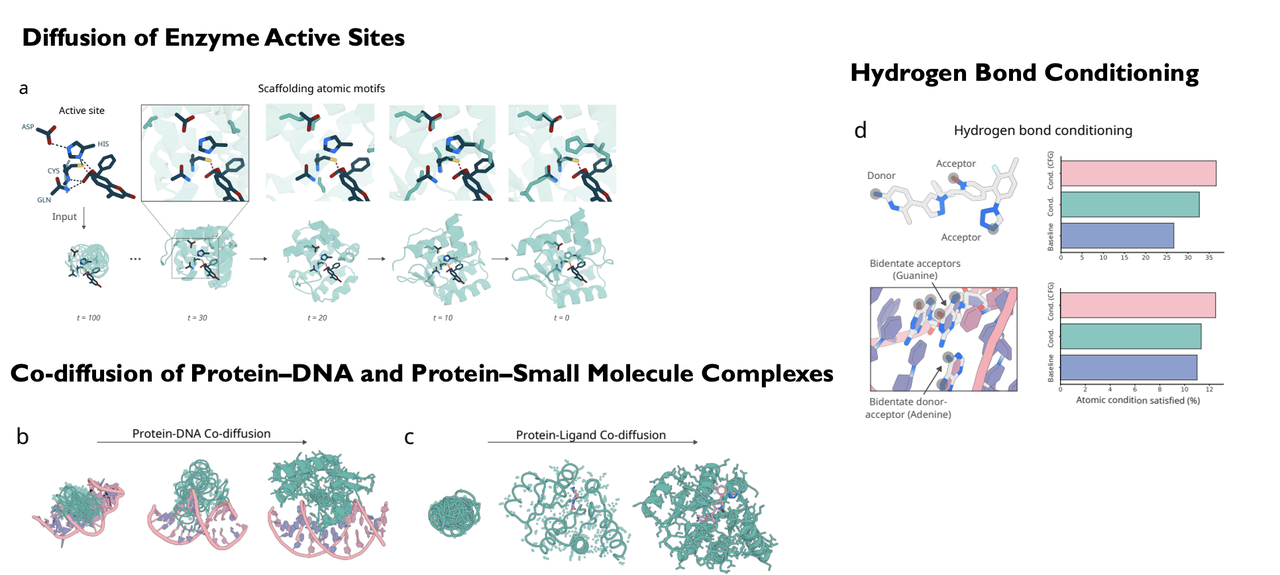
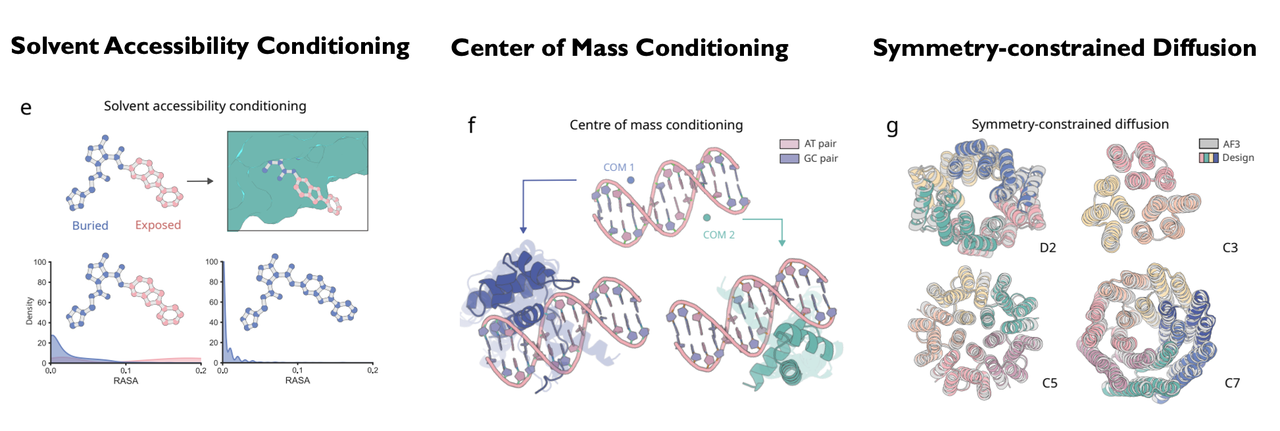
Conditional Control & Programmability of RF3
2️⃣ 高效与性能并存
采用全新Transformer–U-Net结构(重写全部代码)
速度提升约10倍(相较于RFdiffusion2)
同时在四大任务中全面超越前代模型:
蛋白–蛋白结合
蛋白–DNA结合
蛋白–小分子结合
酶活性位点设计
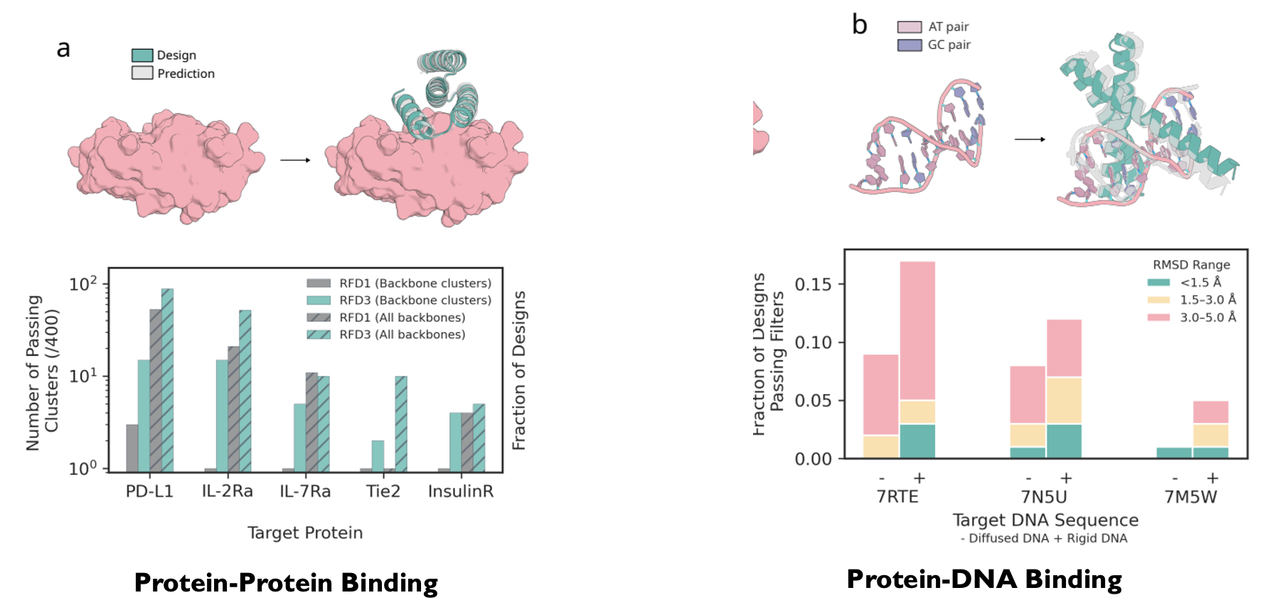
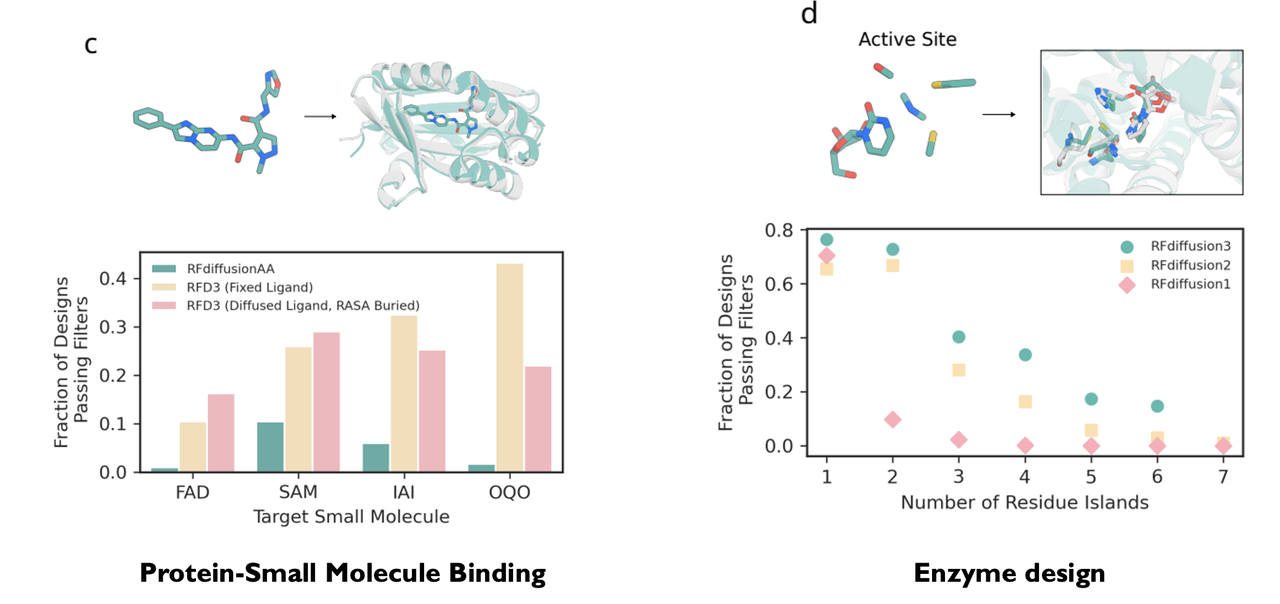
3️⃣ 通用基础模型(Unified Foundation Model)
RFD3首次将所有功能整合进单一架构:
从对称多聚体、酶催化中心,到小分子配体与DNA结合,研究者再也不需要多个独立工具。
这让“通用分子生成模型”的梦想变得现实。
关于RF3的详细内容,也可以看我之前的推送:
实验验证:
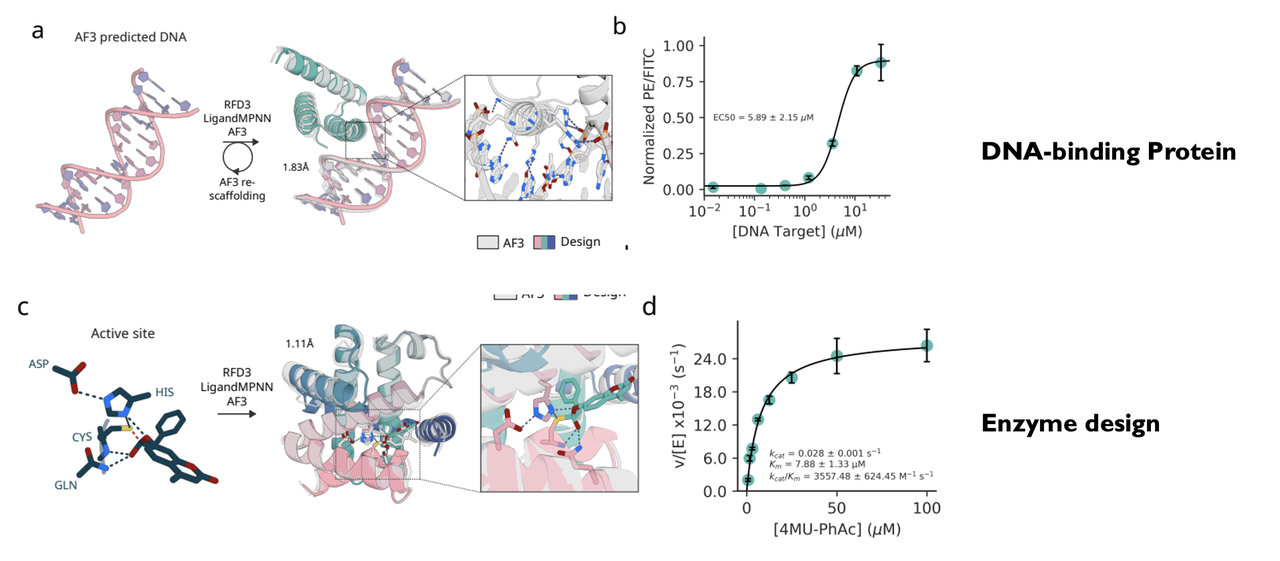
DNA结合蛋白:
模型共生成5种设计,其中一个在实验中展现出
微摩尔级结合力(EC50≈5.9 μM)。
酶设计:
成功设计出可重复催化的半胱氨酸水解酶(Kcat/Km=3557),
活性超过以往所有AI设计的同类酶
开源,共享,重塑科学
RFD3的模型与训练代码已在 Rosetta Commons Foundry (GitHub) 上开源,Github库的链接为https://github.com/RosettaCommons/foundry。

这意味着全球科研人员——无论在美国、中国还是南非——都能使用同一份代码,去训练、改进、扩展这个模型。
“当全世界的实验室都能自由地下载并改进这套代码时,
科学进步的速度会被成倍放大。”
—— David Baker, PhD,IPD主任
快速上手指南:
本地安装指导:
下载代码库:
创建conda环境:
安装:
下载模型权重,可以使用以下命令将所有模型下载到目标文件夹:
这条命令会下载所有已支持的模型(包括 RF3 的多个检查点),
但如果你是初学者,可以先从以下命令开始:
请参阅 examples/all.ipynb,了解如何在笔记本(notebook)中运行每个模型
Google Colab(新手推荐)
若想通过交互式的 Google Colab 笔记本了解如何使用 RFD3、MPNN 和 RF3 进行基础的蛋白设计流程,请参阅 IPD Design Pipeline 教程。
主要包含以下三个主要模块:
欢迎大家分享自己的使用体验,我也要去试试全新开源的RFdiffusion3!(感觉像是喜欢玩的游戏突然更新了)
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →