Nature Biotechnology|PepMLM 实现纯序列驱动的结合肽设计与实验验证
蓝极说:
在蛋白质设计的世界里,有一个长期存在的难题:很多致病蛋白没有稳定的三维结构,让药物分子无处下手。
这类靶点被称作“难成药”蛋白,它们往往正是癌症、神经退行性疾病甚至新发病毒中的关键驱动因子。
今天我要和大家分享的,是刚刚(2025.08.13)发表在 Nature Biotechnology 的一篇文章:
《Target sequence-conditioned design of peptide binders using masked language modeling》(靶标序列驱动的肽结合物设计)。
这篇文章的亮点在于:
它提出了一种全新的思路:完全不依赖蛋白质结构,仅凭目标序列,就能设计出能与其结合的肽。
作者把这种方法命名为 PepMLM,通过语言模型,让模型“读懂”蛋白的语言,然后自己补全出能结合的肽。
更重要的是,这不只是理论模型:作者还在癌症、亨廷顿舞蹈症、以及高致死率病毒上做了实验验证,效果非常亮眼。
也就是说,这篇文章展示了一种可能改变药物研发范式的新方法:
从“结构驱动”到“序列驱动”,让蛋白质语言模型直接生成药物候选肽。

🔗原文链接:https://www.nature.com/articles/s41587-025-02761-2
💻Github链接:https://github.com/programmablebio/pepmlm
一、引言:为什么这项工作重要?
在药物研发里,一个最关键的任务就是找到“结合物”——能牢牢黏住目标蛋白的小分子或者肽段。
小分子药物在过去几十年里成就斐然,但它们往往只能作用于有明确口袋或者沟槽的蛋白。很多“难成药”(undruggable)的靶点,比如转录因子、融合蛋白,根本没有现成的结合口袋,这让设计有效药物非常困难。
近年来,结构预测工具(如 AlphaFold) 的出现,为蛋白设计带来了巨大推动。基于结构的方法,比如 RFdiffusion,可以帮助设计新的结合蛋白。但问题是:
👉 这些方法仍然过度依赖目标蛋白的三维结构。
👉 而实际上,很多致病蛋白是无序的(disordered),没有稳定结构可参考。
于是,这篇文章的作者提出了一个很新颖的想法:
能不能像自然语言处理那样,不依赖三维结构,只基于“蛋白质的语言”(序列),直接让模型去“写”出合适的结合肽?
这就是 PepMLM 的核心:
一个基于 语言模型的肽设计框架,只需要输入目标蛋白的序列,就能设计出与之结合的全新肽分子。
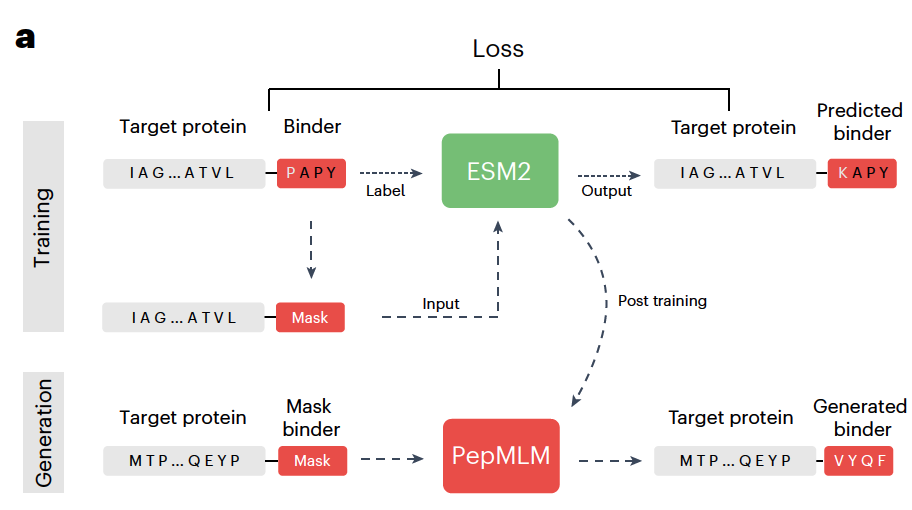
二、方法亮点:PepMLM是如何工作的?
我们知道,语言模型的基本思路是——给它一个句子,把中间挖掉一些词,然后让模型去预测这些缺失的词。
PepMLM 就是把这个思路搬到蛋白质世界:
👉 输入:目标蛋白的氨基酸序列 + 一个被 mask 掉的肽段位置。
👉 输出:模型预测的 结合肽序列。
这样,模型就像在“补全句子”一样,学会如何生成能贴合目标蛋白的肽段。
核心指标:Perplexity (PPL)
文章引入了一个关键概念——困惑度(perplexity, PPL)。
简单理解:PPL 越低,说明模型对这个肽越“自信”,认为它越可能是真正的结合物。
作者把 PepMLM 的生成结果和真实已知的结合肽做比较,发现两者的 PPL 分布几乎重叠,说明模型能很好地模仿真实结合模式。
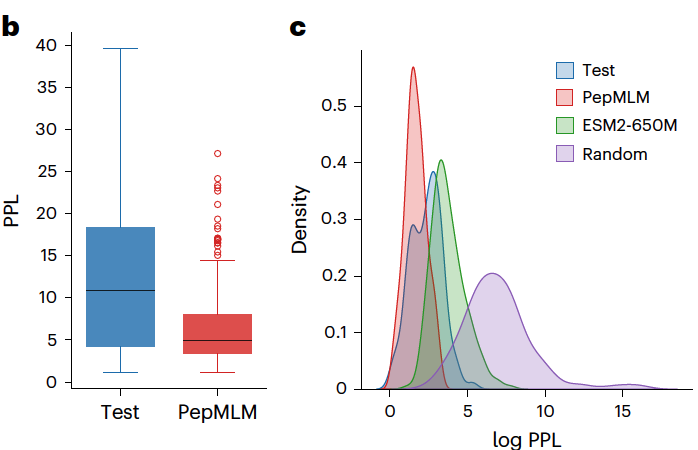
Fig.1b 展示了不同方法生成肽的困惑度分布,可以看到 PepMLM 生成的肽分布(红色)几乎贴合真实肽(蓝色)。
Fig.1c 则进一步证明 PepMLM 能明显区分“真”结合肽和“随机肽”,而原始ESM-2模型和随机序列就做不到。
换句话说,PepMLM 学会了“哪些肽是合理的结合物,哪些只是噪声”。
三、性能对比:PepMLM vs. 现有方法
设计结合肽的挑战,不仅仅是“能生成”,更重要的是:
👉 它是否真的能和目标蛋白牢固结合?
👉 在和现有最强的结构驱动方法(比如 RFdiffusion)相比时,谁更强?
作者用了一系列对比实验来回答这个问题。
- PepMLM 的命中率更高
他们用 AlphaFold-Multimer 来预测 PepMLM 设计的肽是否能和目标蛋白稳定结合,并用 ipTM 分数来衡量结合可信度。
结果显示:
PepMLM 的命中率达到 38%,而 RFdiffusion 只有 29%。
如果进一步要求结合结构的可靠性更高(pLDDT > 0.8),PepMLM 依然保持优势(49% vs. 34%)。
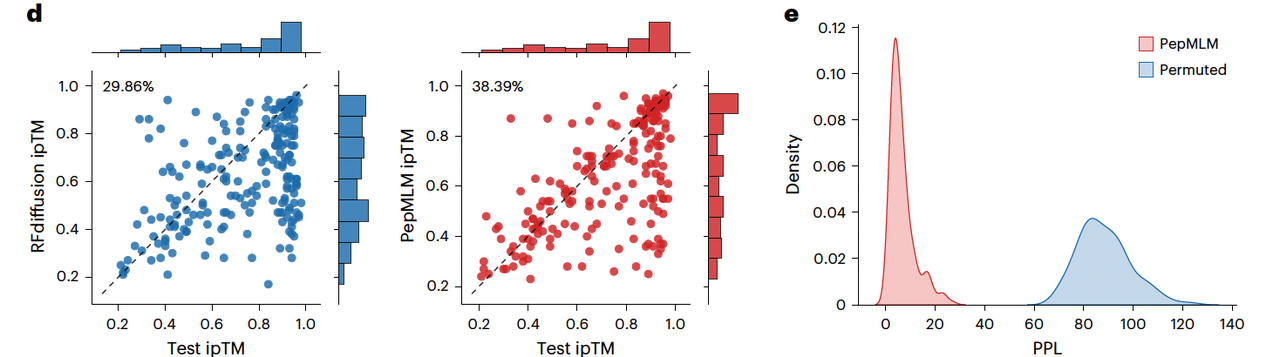
Fig.1d 左右两图直接对比了 RFdiffusion 和 PepMLM 的命中率差异,直观展现了 PepMLM 的性能优势。
- 结合特异性验证
为了确认 PepMLM 并不是“随便匹配”,作者做了一个 permutation test(打乱配对测试):
如果把设计的肽随机分配到错误的蛋白上,PPL 分数明显升高。
说明 PepMLM 确实能识别“对的配对”,而不是生成一些泛化的粘性序列。
Fig.1e 蓝色曲线(正确配对)和红色曲线(错误配对)差异显著(P < 0.001),说明 PepMLM 生成的肽具备 特异性。
- 结构层面的对比
作者还选取了两个具体例子:
小鼠 MHC 蛋白(PDB: 2OI9):PepMLM 生成的肽虽然序列不同,但结合方式和已知肽几乎一致。
人类酪氨酸激酶(PDB: 1LCK):PepMLM 生成的肽结合得甚至比已知肽更牢固(ipTM: 0.82 vs. 0.68)。
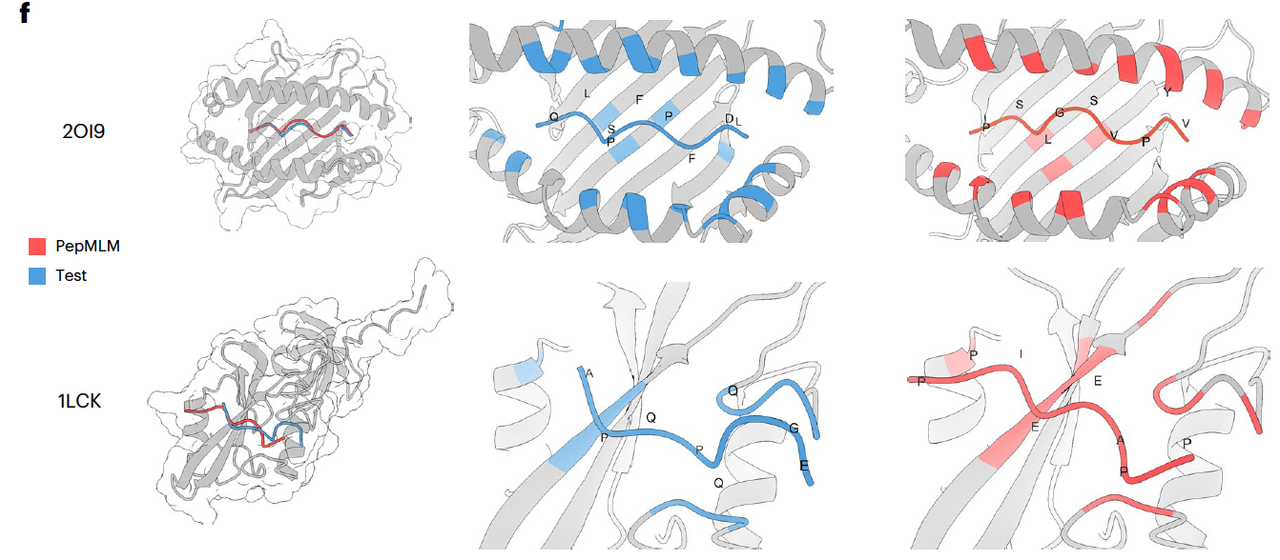
Fig.1f 直观展示了实验肽(蓝色)和 PepMLM 生成肽(红色)在蛋白上的结合结构,非常接近,说明 PepMLM 真的“学会”了结合模式。
总结来看:
PepMLM 不仅在 量化指标(命中率、PPL) 上优于 RFdiffusion,还在 结构层面 展现出高度合理性。换句话说,它是一个 既懂统计分布,又能落地到空间结构 的方法。
四、实验验证:从序列到真实结合
前面我们看到,PepMLM 在计算机模拟里表现很强。但科学界一向谨慎:
💡 模拟再好,也要拿实验说话。
于是,作者在三个层面做了验证:癌症/生殖疾病 → 神经退行性疾病 → 病毒感染。
- 癌症与生殖疾病靶点
作者首先测试了两个重要的受体:
NCAM1:急性髓系白血病(AML)的标志物。
AMHR2:多囊卵巢综合征(PCOS)相关的关键受体。
实验方法:ELISA(检测肽与蛋白结合能力)。
结果:
PepMLM 生成的肽在 纳摩尔级浓度 就能检测到结合信号。
与 RFdiffusion 相比,PepMLM 的成功率和信号都明显更高。
而且这些肽不和 BSA 等无关蛋白结合,说明特异性强。
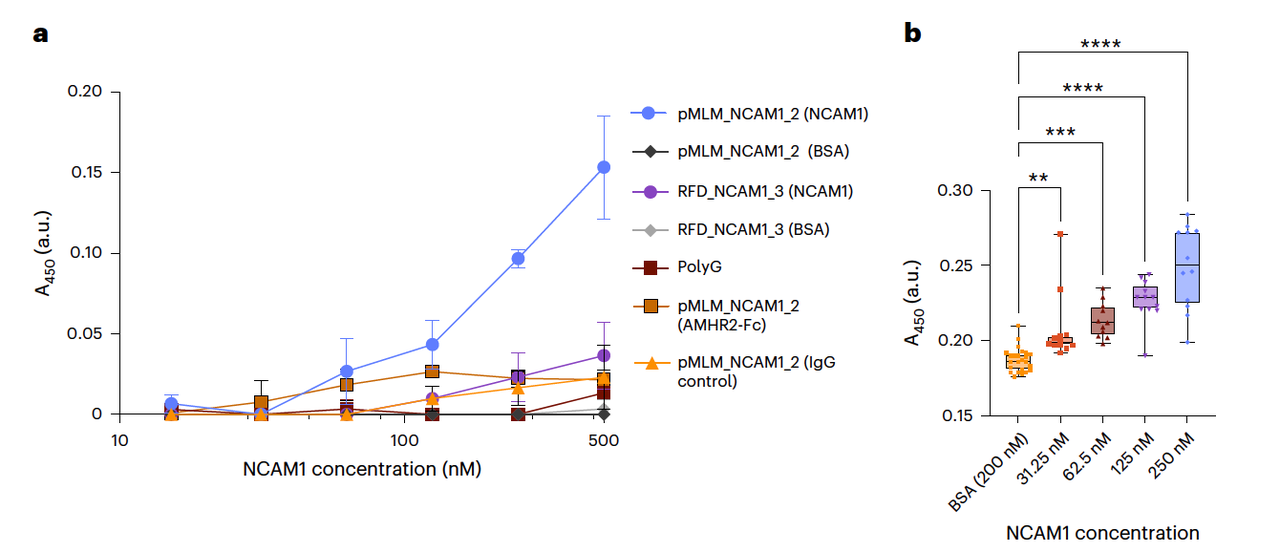
Fig.2a–b 展示了 ELISA 曲线,黑色对照线几乎无信号,而 PepMLM 生成的肽能在低浓度下就拉开差距。
- 亨廷顿舞蹈症(Huntington’s disease)
这是一种因 Huntingtin 蛋白突变 引起的神经退行性疾病。
作者尝试把 PepMLM 生成的肽融合到 uAb(合成泛素连接酶) 上,让它们去标记病变蛋白进行降解。
实验结果:
PepMLM 生成的肽能有效结合并引导降解 MSH3(与疾病进展相关的蛋白)。
更惊喜的是,它们还成功降解了突变的 Huntingtin 蛋白,在细胞里显著降低其水平。
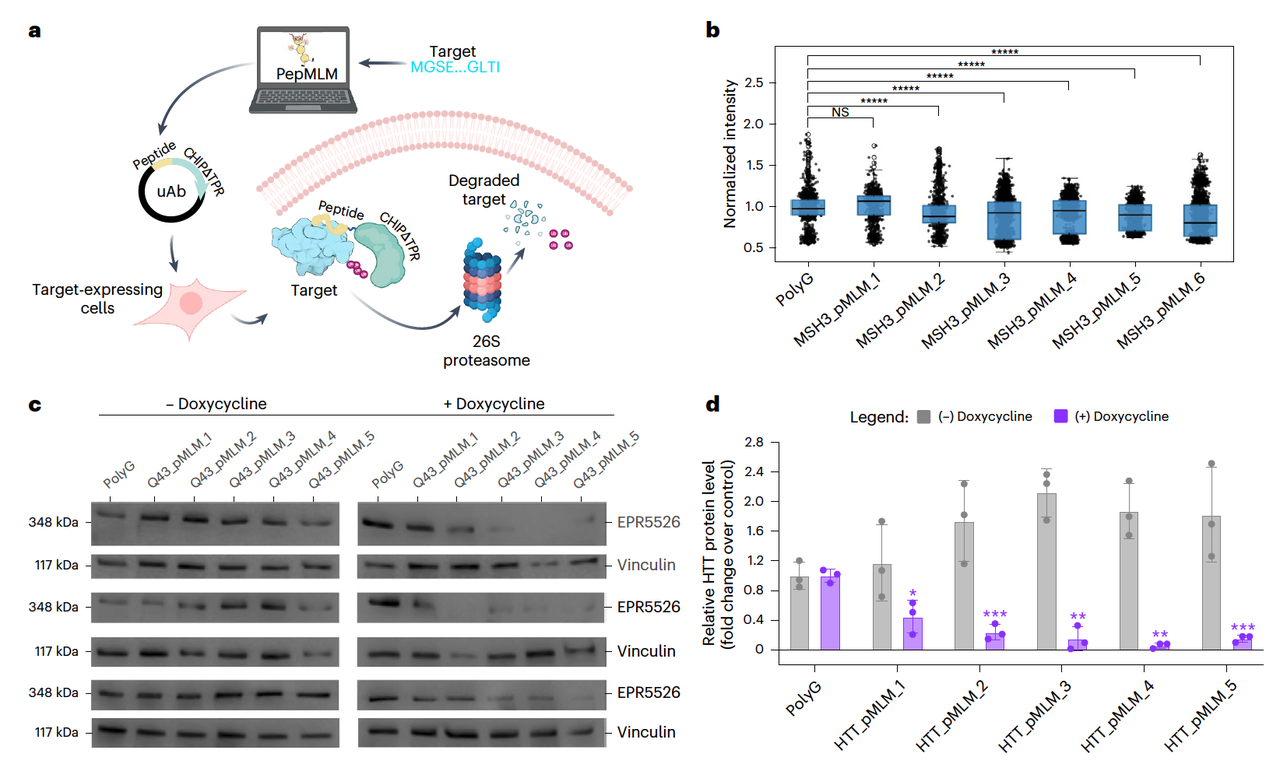
Fig.3b–d 的免疫荧光和 Western blot 清楚展示了:在诱导表达后,Huntingtin 蛋白水平明显下降,PepMLM 肽确实起了作用。
- 病毒蛋白降解
最后,作者把 PepMLM 用到 新发病毒 的关键蛋白上。
选择的靶点是 病毒磷蛋白(P protein),它在病毒复制里非常重要。
测试的病毒包括:
尼帕病毒(NiV)
亨德拉病毒(HeV)
人偏肺病毒(HMPV)
实验结果:
PepMLM 生成的肽引导的 uAbs 能在细胞中显著降低病毒磷蛋白水平。
特别是在 HMPV 感染实验里,免疫荧光成像显示病毒蛋白几乎被“清空”。
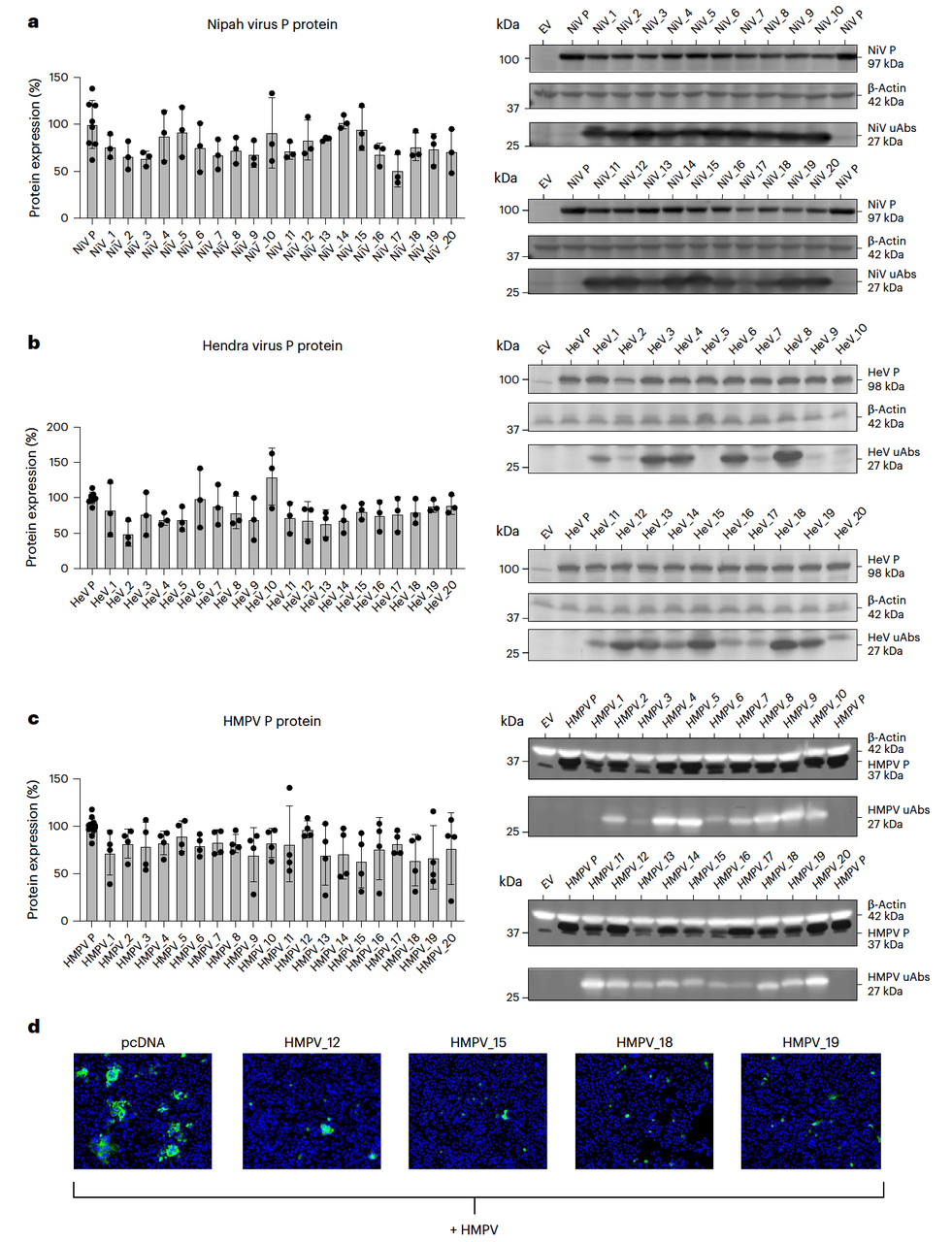
Fig.4a–d 展示了 Western blot 的条带和免疫荧光图,绿色信号(病毒蛋白)在 PepMLM 处理组里明显减弱甚至消失。
小结
通过 体外结合实验、细胞蛋白降解实验,再到 病毒感染实验,作者证明了:
👉 PepMLM 生成的肽不仅能“理论上结合”,而且能在真实的生物系统里 高效、特异性地结合并调控目标蛋白。
这一步让 PepMLM 从“一个AI算法”真正跨入了“潜在药物研发工具”的行列。
五、意义与展望
通过前面的实验可以看到,PepMLM 的价值远不止“能生成肽”。它实际上开辟了一条全新的药物设计路径:
- 从“结构驱动”到“序列驱动”
过去的蛋白设计方法往往依赖 三维结构,比如用 AlphaFold 预测结构,再基于结构建模结合位点。
但 PepMLM 证明了:
👉 仅仅依赖目标序列,模型也能学会生成合理的结合肽。
这让很多缺乏稳定结构的“难成药”靶点变得可攻击,比如转录因子、无序蛋白等。
- 应用范围的广阔性
文章已经展示了几个应用方向:
肿瘤标志物与生殖疾病靶点(NCAM1、AMHR2);
神经退行性疾病(亨廷顿舞蹈症相关蛋白 MSH3、突变 Huntingtin);
新发病毒的关键蛋白(尼帕、亨德拉、人偏肺病毒的磷蛋白)。
未来,任何只要知道序列的蛋白,都可能成为 PepMLM 的设计对象。
换句话说:它为 “通用的、序列驱动的结合肽生成器” 奠定了基础。
- 对药物研发的启发
PepMLM 不仅能生成肽,还能作为 降解剂(uAb 引导) 的模块,帮助特异性地清除致病蛋白。
这意味着:
在癌症治疗中,它可以针对耐药蛋白或异常活化的通路蛋白。
在神经疾病中,它能靶向难以用小分子抑制的聚集蛋白。
在传染病中,它能快速响应新发病毒,直接从序列出发设计抗病毒肽。
未来,随着模型与实验闭环(比如高通量验证结合 AI 优化),PepMLM 有潜力成为一个 “CRISPR for proteins” ——像 CRISPR 对 DNA 那样,让研究者可以编程化地控制蛋白质的命运。
- 展望
作者也提到了一些后续改进方向:
加入 修饰信息(比如翻译后修饰、变体信息),让设计更贴近真实生物环境;
结合 环化、stapling 等肽修饰策略,提高药物稳定性;
与 高通量实验平台 联合,形成“AI-实验反馈”的闭环,加速优化。
这些方向意味着,PepMLM 不仅是一个科研工具,还可能成长为 药企和合成生物学公司实际可用的平台。
小结
PepMLM 的最大意义在于,它证明了:
👉 理解蛋白质的“语言”就足以设计结合分子。
这是一个从语言建模走向药物研发的新范式。
它让我们看到,未来针对癌症、神经疾病乃至新发传染病,我们可能不再被“结构限制”,而是可以直接从序列出发,快速获得潜在药物候选。
六、总结
在这篇文章中,作者提出了 PepMLM ——一个基于语言模型的靶标序列驱动肽设计方法。
它的亮点在于:
只需序列,无需结构,也能生成特异性的结合肽;
在 体外实验和细胞实验 中都表现优异,能有效结合甚至降解疾病相关蛋白;
应用范围跨越 癌症、神经退行性疾病到病毒感染,展示出通用性和前景。
如果说以往的药物设计像是在黑暗中摸索结构口袋,那么 PepMLM 更像是直接“读懂”了蛋白质的语言,从语言中生发出结合策略。
它的意义不止在科研层面,更可能成为未来 药物研发的新范式 ——一个真正的“序列即药物”平台。
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →