PepCCD:用对比学习与扩散模型实现序列驱动的多肽设计
今天想和大家分享一篇最新的工作 —— 《PepCCD: A Contrastive Conditioned Diffusion Framework for Target-Specific Peptide Generation》。

为什么要讲这篇文章呢?
一方面,多肽药物在“不可药物化”靶点上的潜力越来越受到关注,但现有的设计方法不是过度依赖高分辨率结构,就是需要昂贵低效的实验筛选。
另一方面,这篇文章提出的 PepCCD 框架,把 对比学习 和 扩散模型 融合到了一起,实现了仅基于蛋白质序列的端到端多肽生成。这种方法论上的突破,可能会成为未来肽药设计的一个新范式。
对我来说,这篇文章最大的亮点在于:它不仅解决了“没有结构怎么办”的难题,还在多样性、效率和生物活性预测上超越了已有的基于序列的方法。可以说,它为“序列驱动的肽药设计”打开了一扇新的大门。
第一部分:背景介绍
在现代药物研发中,有一个长期存在的难题:超过 80% 的致病蛋白被认为是“小分子不可药物化”的靶点,因为它们缺乏稳定的结合口袋,难以被传统的小分子抑制剂有效调控。这类靶点,往往是疾病进程的关键角色,却常常被挡在“药物化”大门之外。
相比之下,多肽药物以其高特异性、良好生物相容性和低毒性,在这类靶点的治疗中展现出巨大潜力。过去二十年,已有超过百种多肽药物成功上市,用于癌症、糖尿病、感染性疾病等多种领域。但问题也随之而来:
如何在庞大的多肽序列空间里,高效找到真正能与特定蛋白结合的候选?
传统路径依赖于 高通量实验筛选 或 基于蛋白结构的计算设计。然而,实验筛选不仅耗时耗资,成功率也低;而结构依赖的方法,又受限于高分辨率结构数据的稀缺性和不稳定性,特别是面对动态构象蛋白时,几乎无能为力。
那么,能否只依靠蛋白质的序列信息,就直接生成与之特异结合的多肽?
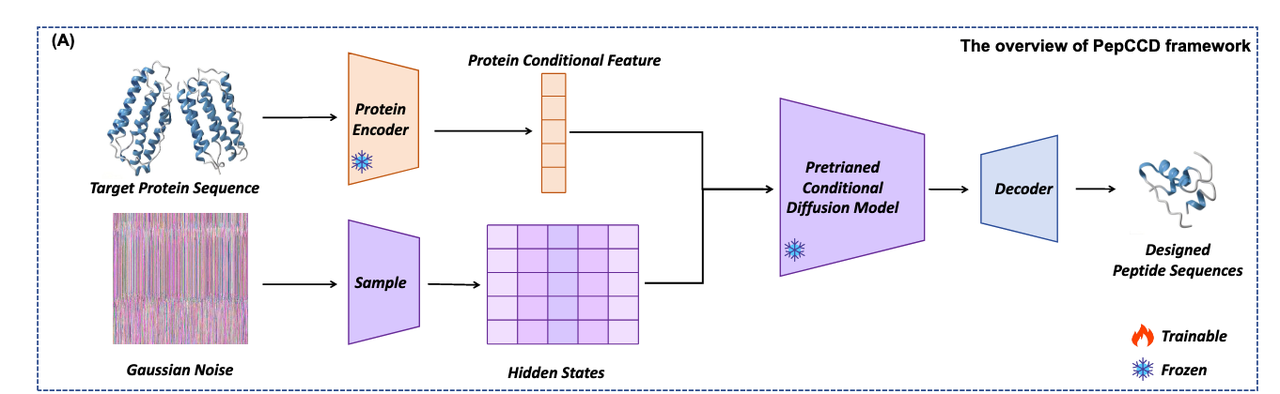
这一问题,正是本文所要讲解的研究——PepCCD 框架——试图回答的核心。正如 Figure 1(A) 所展示的那样,研究者们设计了一个端到端的生成式模型,只需输入蛋白序列,就能输出具有靶点特异性的候选多肽,彻底绕开对结构的依赖。
第二部分:方法创新逻辑
PepCCD 的设计框架可以从 Figure 1 来理解:从输入蛋白质序列开始,经过三个核心阶段,最终输出与目标蛋白高度匹配的多肽序列。相比于以往依赖结构的设计流程,这一方法的最大创新,在于它把 对比学习(Contrastive Learning) 和 条件扩散模型(Conditional Diffusion Model) 有机结合,完成了“仅基于序列”的端到端多肽生成。
- 框架总览(Figure 1A)
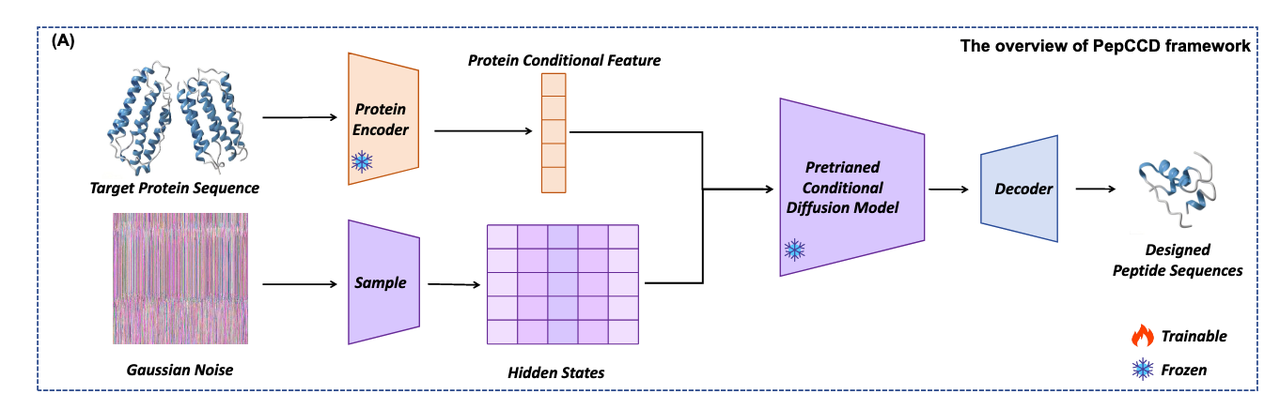
研究者首先构建了一个整体的Pipeline:输入是蛋白质序列,输出是目标特异性多肽序列。整个过程完全不依赖三维结构,只需要序列信息,就能走通完整的生成路径。这为“结构不可得”的靶点,提供了全新的设计可能。
- Stage 1: 蛋白-多肽对比学习 (Figure 1B)
核心问题在于:
怎样让模型理解“这个蛋白应该匹配什么样的多肽”?
PepCCD 的解决方案是引入对比学习。
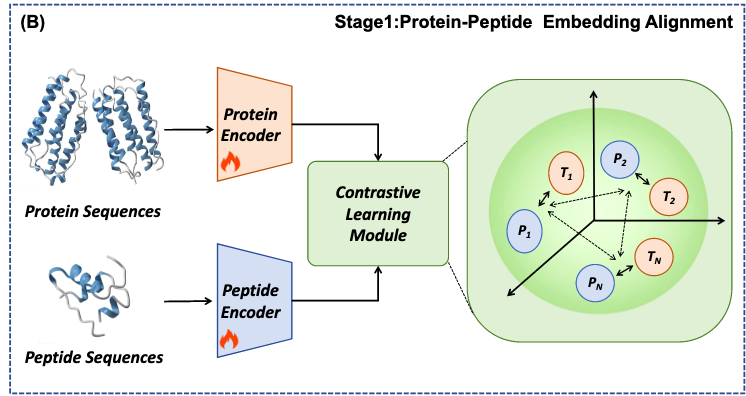
使用双 ESM-2 编码器,分别对蛋白和多肽进行编码。
通过对比学习,把真实配对的蛋白-多肽 embedding 拉近,把非配对的推远。
最终在语义空间中,蛋白序列就能携带关于“潜在结合肽”的关键信息。
这一阶段的意义在于:让蛋白和多肽共享语义空间,为后续的条件生成提供“桥梁”。
- Stage 2: 无条件的多肽扩散模型预训练 (Figure 1C)
接下来,研究者构建了一个超大规模的模拟多肽数据集,对扩散模型进行 无条件预训练。
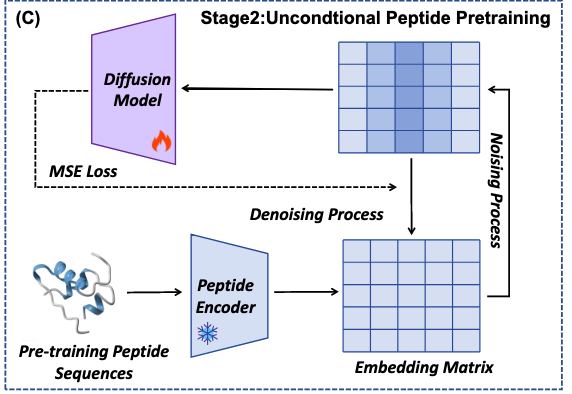
过程如图所示:对多肽 embedding 加入高斯噪声,再学习去噪还原。
本质上是让模型掌握多肽序列的整体分布和多样性。
这一阶段的意义在于:为模型打下“多肽长什么样”的统计学基础,保证后续生成不至于局限在模板或训练数据里。
- Stage 3: 条件扩散微调 (Figure 1D)
最后,PepCCD 将 目标蛋白的语义向量(来自 Stage 1 对比学习)输入扩散模型,作为条件信号,引导去噪过程。
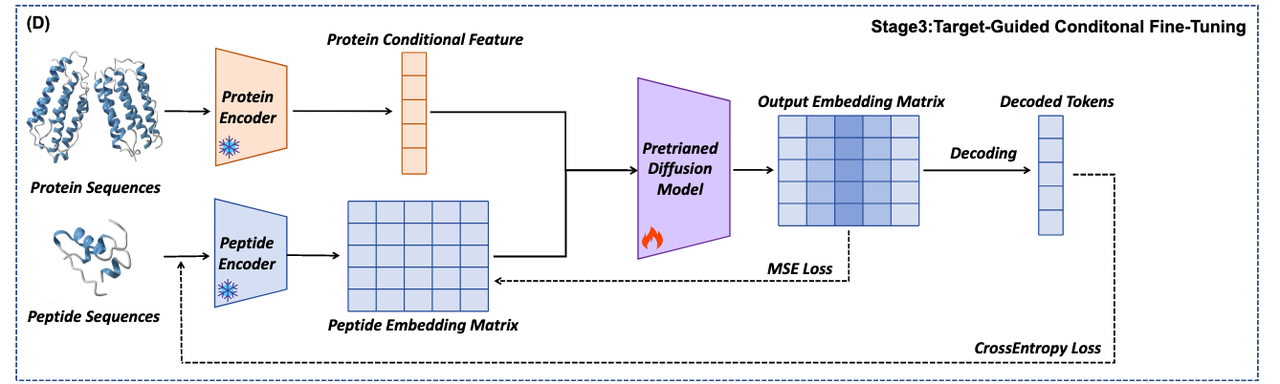
在这一环节,生成过程不仅能保持多肽的多样性,还能精准对齐到特定蛋白靶点。
输出阶段,模型会解码成具体的氨基酸序列,并通过交叉熵损失优化与真实数据的接近度。
总结一下:
Figure 1 展示的三阶段流程,正是 PepCCD 的创新核心:
Stage 1 确保蛋白-多肽的“语义桥梁”;
Stage 2 提供多肽生成的多样性与稳健性;
Stage 3 则把二者结合起来,实现精准且多样的靶点特异性设计。
第三部分:实验结果亮点
模型提出后,研究者将 PepCCD 与两类最先进方法做了对比:
RFdiffusion:基于结构的代表方法;
PepPrCLIP:基于序列的代表方法。
对比结果直接体现在 Table 1、Table 2 和 Figure 2 里。
- 更强的靶点结合能力(Table 1)

在 ipTM 和 Rosetta 总能量分数 等衡量蛋白-多肽复合物结合力和稳定性的指标上,PepCCD 明显优于传统的序列引导方法。
虽然在某些结构信息极其有利的情况下,PepCCD 略逊于 RFdiffusion,但这是因为 RFdiffusion 直接利用了三维结构,而 PepCCD 完全不依赖结构。
换句话说,PepCCD 证明了:即使只有序列输入,也能设计出高质量的多肽。
- 更高的多样性与生物活性(Table 2)
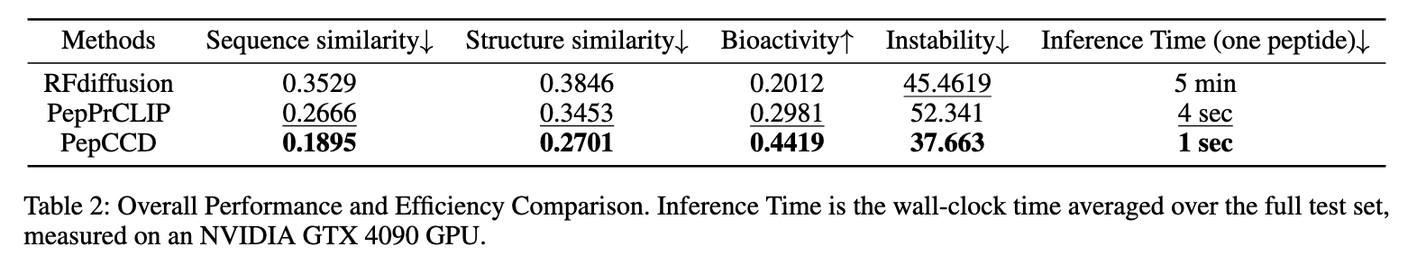
PepCCD 在 序列相似度 和 结构相似度 两项指标上最低,说明生成的候选肽最具新颖性和多样性。
在 生物活性预测分数 上,PepCCD 也显著高于其他方法,这意味着它更有可能生成具有功能性的候选分子。
另外,PepCCD 生成的多肽在 稳定性 上更好,减少了体外实验失败的风险。
这些结果共同说明:PepCCD 不仅生成“能结合”的肽,还生成“多样且可用”的肽。
极致的推理效率(Table 2)
PepCCD 平均生成一个多肽只需 1 秒,而 RFdiffusion 需要 5 分钟。
这种数量级上的差距,让 PepCCD 在大规模虚拟筛选中展现出压倒性优势。
- 命中率的直观对比(Figure 2)
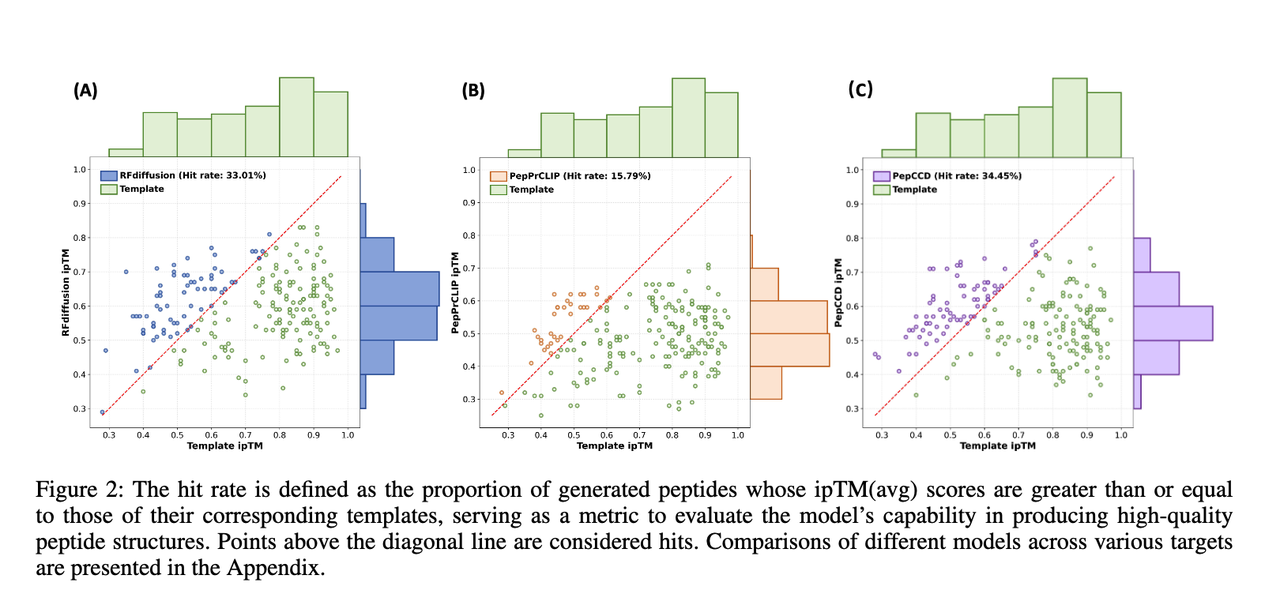
Figure 2 把不同方法的 命中率 画在对角线坐标上:点落在对角线以上,说明生成的肽比参考模板更优。
结果显示,PepCCD 在绝大多数靶点上都能“超越模板”,并且在平均水平上甚至超过了基于结构的 RFdiffusion。
这意味着:PepCCD 在没有结构信息的条件下,仍能稳定地输出高质量候选。
小结
PepCCD 的实验结果非常亮眼:
在靶点结合力、多样性和生物活性上全面领先;
在推理速度上远超竞争对手;
在实际命中率上甚至可以比肩或超越结构驱动方法。
它展示出了一种真正可扩展、可实用的“仅依赖序列”的多肽设计路径。
第四部分:关键创新总结
读到这里,不难发现 PepCCD 的独特性:它并不是对现有方法的“小修小补”,而是在方法论上实现了一个真正的突破。
- 首次引入扩散模型做多肽设计
过去扩散模型多应用于图像、蛋白结构生成,PepCCD 则把它引入到多肽序列生成中。
这使得模型能够在庞大序列空间中高效探索,生成结果既多样又稳定。
蛋白-多肽对比学习的创新应用
对比学习在 NLP 和计算机视觉中已成熟,但应用于蛋白-多肽关系还是第一次。
它让蛋白序列本身就能携带关于“潜在结合肽”的信息,从而实现真正的 序列驱动设计。
- 三阶段训练范式
对比学习(Stage 1):建立语义桥梁;
无条件预训练(Stage 2):学会多肽的多样性和分布;
条件微调(Stage 3):把蛋白条件和扩散生成结合,输出目标特异性候选。
三者结合,使 PepCCD 同时具备 特异性 和 多样性,避免了“只会生成一个模板化肽”的局限。
- 摆脱对结构的依赖
传统方法要么靠高分辨率结构,要么依赖模板序列。PepCCD 的核心价值在于:仅凭蛋白序列,就能设计出高质量的多肽。
这对动态构象蛋白、无结构数据的靶点尤其重要。
PepCCD 的最大创新,在于用对比学习建立蛋白-多肽语义桥梁,再通过条件扩散模型实现端到端的序列驱动生成,从而真正突破了“无结构不设计”的限制。
第五部分:蓝极的思考
PepCCD 展示了一条全新的路径:仅依赖蛋白序列信息,就能生成多样且特异性的多肽。这一点,尤其对那些缺乏高分辨率结构数据的“难缠靶点”,具有非凡意义。
不过,我们也需要看到这项工作的局限性:
缺乏湿实验验证:目前的评估主要依赖计算指标(如 ipTM、Rosetta 分数、分子动力学模拟等)。虽然这些指标在一定程度上能反映结合能力和稳定性,但最终多肽是否真的能在细胞或动物模型中发挥作用,还需要通过实验验证。
数据依赖性:PepCCD 的对比学习依赖于蛋白-多肽复合物数据集,而这些数据在真实世界中仍然有限,可能影响模型在极端冷门靶点上的泛化能力。
换句话说,PepCCD 在 算法层面 有突破,但从 药物开发走向临床,还有一段路要走。
展望
如果后续结合湿实验筛选,验证 PepCCD 生成的候选肽是否真的具备良好的药效和安全性,它将有望成为 肽药设计的核心工具。
更长远地看,这种“序列驱动 + 生成模型”的思路,也许还能扩展到 蛋白-小分子、蛋白-抗体 等更多场景。
所以问题来了:
你觉得序列驱动的方法,未来能不能完全取代依赖结构的方法?还是说它们更可能是互补的关系?
欢迎在评论区留下你的看法,一起探讨肽药设计的未来。
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →