Nature Nanotechnology|从头设计可对 pH 响应的自组装螺旋蛋白丝
今天要和大家分享一篇来自华盛顿大学(University of Washington)蛋白设计研究所(Institute for Protein Design, IPD)的文章,发表在 Nature Nanotechnology(2024 年 7 月刊)上,题为:
《De novo design of pH-responsive self-assembling helical protein filaments》
(“从头设计可对 pH 响应的自组装螺旋蛋白丝”)

这篇论文的通讯作者是蛋白设计领域的标志性人物 David Baker 教授,第一作者为 沈浩(Hao Shen)博士 —— 他是 Baker 团队的重要成员,长期从事“蛋白质自组装体系的计算设计”。这项工作由华盛顿大学、剑桥大学、以及太平洋西北国家实验室多方合作完成。
这篇文章的核心问题是:
“我们能否用计算机设计出一类能感知环境变化、并像天然材料一样自发重构的蛋白质结构?”
简单来说,研究团队成功地 从头设计(de novo design)了一种在 pH 改变时能自行组装或解组的螺旋蛋白纤维。而且这种响应极为灵敏——仅仅 0.3 个 pH 单位的变化,就能让纤维在几百毫秒内“瞬间消失”;当 pH 恢复时,它们又会重新长回去。
在接下来的内容中,我们将带大家一起从图文角度出发,看看 Baker 团队是如何一步步实现这个看似“自然界才会做的事”:
他们如何在计算机中设计这种 pH 响应的自组装单元;
如何通过 冷冻电镜(Cryo-EM)验证原子级精度的结构一致性;
以及如何用显微镜和 AFM 动态观察 纳米级“丝的生与灭”。
一、从自然界的智慧到计算机的设计
自然界早已在分子层面玩出了“环境响应材料”的巅峰操作。
当蜘蛛在吐丝时,丝蛋白在腺体内会经历一个从中性到酸性的微小 pH 变化——这种变化就像一个分子开关,使蛋白质链自发组装成坚韧的丝纤维。
在酵母中,CTP 合酶在低 pH 条件下聚合成丝状结构,帮助细胞在饥饿时维持代谢平衡;而某些细菌的 R bodies,则能在酸性环境下“伸展爆裂”,突破宿主膜释放物质。
这些例子共同说明了一件事:pH 变化可以被生物系统精准地“编程”成结构变化的信号。
但是,要从零开始设计一种能像天然体系那样精准响应的人工蛋白丝,却非常困难。
天然系统依赖数百万年的演化优化,而人工设计需要计算机在原子层级上同时考虑折叠稳定性、组装几何、以及响应性残基的网络效应——这几乎是“分子工程中的珠穆朗玛峰”。
1. Baker 团队的构想:从 pH 响应三聚体出发

研究团队选择以一个已经验证过的 pH 响应三聚体结构(pRO-2.3) 为基础。
这个三聚体内部埋藏着 六个组氨酸(His)残基,它们通过氢键网络稳定整个结构。
当 pH 降低、组氨酸被质子化时,氢键网络被破坏,三聚体就会解体——这正是一个“天然的分子开关”。
在图 1a 的上半部分,可以看到研究者通过添加短 连接肽环(loop) 把这三个亚基连成了一个单链结构单元(monomer)。
这样一来,他们打破了原本的三重对称性,为后续设计“非对称界面”创造了空间。
而在图 1a 的下半部分标出了那些关键的氢键网络——其中每个组氨酸都像一个敏感的触点,决定着结构何时会“开”与“关”。
2. 从单体到纤维:计算设计的螺旋装配
接下来,研究团队利用 Rosetta 设计平台,探索了 45,000 种可能的螺旋堆叠方式。
他们像搭积木一样,让这些单体沿着不同的旋转角度与上升距离组合成各种螺旋模型(Fig. 1b)。
每种构型都经过能量评估、形状互补性和界面面积的筛选,最终选出了 18 种能量最低、理论上最稳定的方案。
在图 1c 中,每个亚基用不同颜色的棒状螺旋表示,之间的红色棒状结构就是 pH 敏感的氢键网络。
这些“带有敏感神经的模块”堆叠在一起,就构成了一根可以对环境微小变化迅速响应的蛋白质丝。
最终,18 种候选设计在 E. coli 中表达后,只有两个设计成功形成丝状结构——DpHF7 与 DpHF18。
这意味着,计算预测的能量最低模型,确实能在实验中自组装成纤维,验证了设计逻辑的可行性。
二、从计算机到显微镜:原子级别的“精准复刻”
当设计完成后,最关键的问题是:
“计算机画出来的蛋白丝,真的能在实验中按照那个形状自组装吗?”
这正是 Fig. 2 要回答的核心问题。研究团队通过 冷冻电镜(Cryo-EM),分别解析了两种设计的结构:
早期版本 DpHF18 和优化后的 DpHF19。
- 结构验证:从模型到真实结构

在 Fig. 2a 的第一列,是计算机预测的设计模型;第二列,是冷冻电镜拍到的纤维照片;第三、第四列分别展示了 DpHF18 和 DpHF19 的三维密度图。
可以清楚地看到:
DpHF18 虽然能形成纤维,但实际装配方式略有偏差;
DpHF19 经过五个关键位点突变后,结构与模型几乎完美重合(界面残基 RMSD 仅 1.4 Å!)。
这意味着,Baker 团队在原子层面实现了结构设计与实验结果的对齐。
这在“从头蛋白设计(de novo design)”领域是极其罕见的成就。
2. 从反平行到完美对称:五个突变的艺术
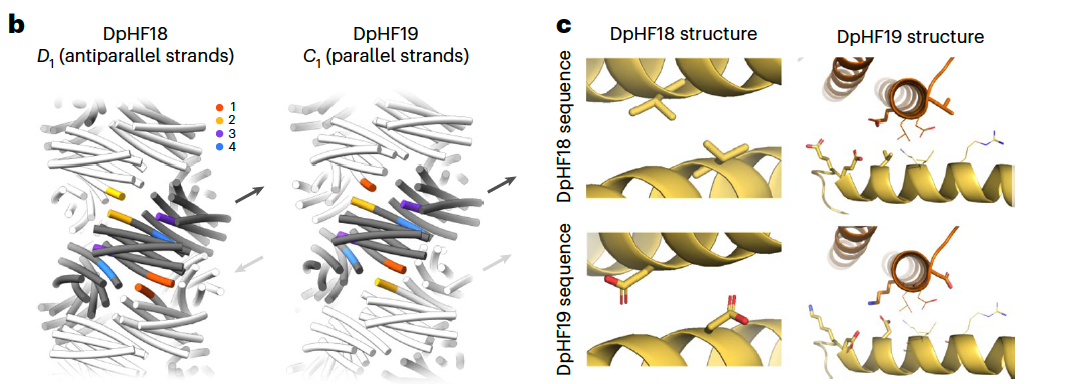
在早期版本 DpHF18 中,纤维的亚基以一种“反平行排列(D1 对称性)”方式堆叠,这会导致纤维内部出现“非设计的界面”,造成结构偏差。
研究团队发现这个问题后,通过对界面残基的精准改造——仅仅五个氨基酸替换,就让结构转变为他们原本设计的“平行螺旋(C1 对称性)”。
在 Fig. 2b 中,左侧是 DpHF18 的反平行结构,右侧是优化后的 DpHF19 的平行结构;方向箭头标出了两者完全不同的对称性。而 Fig. 2c 展示的,就是那几个至关重要的突变位点:两个突变(V29D 和 L82D)破坏了非目标界面,另外三个突变(E32D、E33K、E87K)强化了目标界面的氢键网络。
可以说,这一轮微调让整个体系从“能装”变成了“精准装”。
3. 一致性验证:原子级“重合”

在 Fig. 2d–e 中,研究者将 DpHF19 的 cryo-EM 结构(青色)与计算模型(灰色)叠合,
结果几乎无法分辨差异——这意味着模型中的每一圈旋转、每一个氨基酸朝向、每一个亚基的相对位移,
都与实验观测一致。
这不仅是一次设计成功,更是一次理论可预测性的证明:
“我们可以在计算机中精确地设计出一个会自组装、可响应的蛋白纳米结构。”
三、在 1 秒之间:蛋白丝的“呼吸”
当这些设计好的蛋白丝真正组装起来后,研究团队开始测试它们对 pH 的响应。
结果令人震撼——只要溶液的 pH 略微下降 0.3 个单位,丝就会瞬间解体;而当 pH 恢复,它们又能在几秒内重新组装成形。
就像自然界的丝蛋白在腺体里由液体变为固体,这些“人造丝”也在实验室中展现出同样的灵敏性与可逆性。
1. 分步降 pH:纤维的“呼吸周期”

研究者首先用负染电镜观察了三种设计:
DpHF18、DpHF19 和 DpHF19_9his(后者含 9 个埋藏组氨酸)。他们逐步调节溶液 pH(从 8 降到 6、5、4.2、3.5、3),然后再升回中性(pH 8),观察纤维长度的变化。
从 Fig. 3a 的箱线图中可以看到:
在中性条件(pH 8)下,纤维整齐而延展;
当 pH 降到临界点时,纤维长度骤减至零;
当 pH 再度升高,纤维又重新出现。
更令人印象深刻的是:
不同设计的解装 pH 点不同,
DpHF18 在 pH 3.5 解装;
DpHF19 在 pH 3.0 解装;
DpHF19_9his(9 个组氨酸)在 pH 4.2 解装。
这说明 通过调节组氨酸数量,就能精确“调节响应阈值”。
这在蛋白设计领域是一个极其重要的控制能力。
可以把这幅图理解成分子的“呼吸曲线”:
pH 降低——蛋白丝呼出一口气(解装);pH 回升——它又吸气复位(重组)。
2. 分子显微镜下的“丝的生长”

接下来,团队用 全内反射荧光显微镜(TIRFM) 拍摄了纤维的组装过程。他们将红色荧光(Cy5)标记的“老纤维”与绿色荧光(Oregon488)标记的“新单体”混合,结果看到:绿色的荧光信号只在红色纤维的两端延伸,意味着这些蛋白丝的生长是从“末端”延伸的,就像细胞骨架微管那样有极性。
这说明这些人造丝不仅能组装,还能动态延伸与修复,具备天然蛋白聚合体系才有的方向性装配行为。
3. 毫秒级解装:一个真正的“分子开关”

团队在流动池中让溶液 pH 从 8 快速降低到 3,并实时拍摄 DpHF18–Cy5 纤维的变化。
结果几乎令人不敢相信:不到 1 秒钟,整根纤维就完全解体(见 Fig. 3d)。
在 Fig. 3e 的折线图中可以看到——当 pH 仅从 3.4 降到 3.1,纤维长度曲线瞬间坍塌;而在 3.4 时却依然稳定存在。
换句话说,这个体系对 pH 的响应锐度达到 0.3 个 pH 单位以内,
是一个真正意义上的“分子级数字开关(molecular digital switch)”。
4. 总结:分子层级的灵敏与可逆
这组实验揭示出几个极具启发性的结论:
设计的蛋白丝可在中性 pH 下稳定存在,在酸性下完全解装;
响应过程极快(<1 秒)且完全可逆;
增加埋藏组氨酸的数量,可精确调节开关的触发点;
自组装与解组过程都遵循高度协同性,体现出精细的能量调控。
四、在显微镜下“看见”分子的解体
前一节我们看到了这些蛋白丝能在宏观层面上实现可逆组装。
但 Baker 团队想更进一步——
“这一切到底是怎么发生的?
一根丝,在解体时,会从哪里开始崩解?
解装是瞬间碎裂,还是从端点一点点消融?”
为了回答这些问题,他们使用了液相原子力显微镜(AFM)——一种能够在溶液中实时观察纳米结构表面动态变化的技术。
1. pH 下降时的“融化电影”
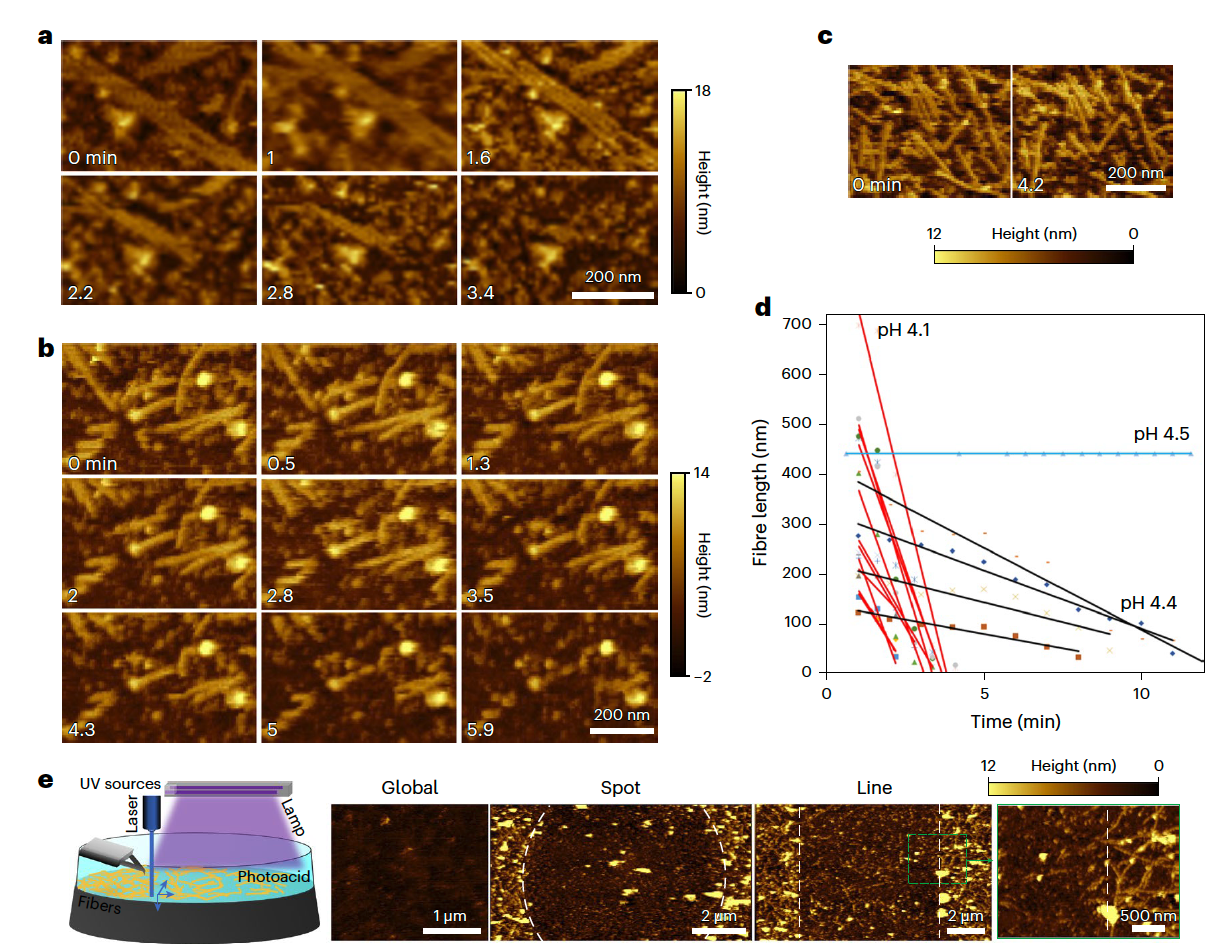
在实验中,研究者将 DpHF19_9his(含 9 个组氨酸)的纤维轻轻铺在云母片上,然后缓缓将溶液 pH 从 8 降到 4.5、4.4、4.1。
接着,他们连续拍摄了每一分钟的形貌变化(如 Fig. 4a–c 所示)。
画面中,我们看到的场景是这样的:
在 pH 4.5 时,丝还纹理清晰、几乎不动;
当降到 pH 4.4,丝的末端开始轻微塌陷,局部变短;
再到 pH 4.1,整条丝快速“融化”消失,仿佛被酸轻轻“抹去”。
AFM 的高度曲线显示,丝的平均长度缩短速率在 pH 4.1 时达到 108 nm/min,而在 4.4 时仅为 21 nm/min。
短短 0.3 个 pH 单位的差异,就能带来五倍的速度变化。
这再次证明:这些丝的响应是高度协同、超灵敏的。
2. 解装机制:从两端开始的“分子消解”
进一步的单丝追踪分析表明:
大多数纤维在两端同时解体,长度对称地缩短;
但也有一些纤维因为一端吸附在表面上,只从另一端“融化”;
有时,纤维会先断裂成几个短段,再从各段两端继续分解。
这些现象说明,丝的解体并非单一模式,而是一种多机制协同过程。
每一根丝都像有“自己的命运”,但总体规律却非常一致。
3. 光控酸触发:用光“写图案”的分子艺术
Baker 团队在这一节做了一个非常酷的实验 ——
他们往溶液里加入一种叫 2-硝基苯甲醛(2-nitrobenzaldehyde) 的光酸分子(photoacid)。
当光酸被紫外光照射后,会释放质子,使局部 pH 降低。
于是他们有了一个大胆的设想:
“如果我们用光照局部区域,能不能只让那一块的丝解体,而其他区域保持完整?”
实验结果堪称艺术品。
如 Fig. 4e 所示:
当用 405 nm 激光点照时,纤维只在照射区域内消失,形成精确的“点”或“线”图案;
当用 364 nm 灯光全局照射时,整个样品表面的纤维都被同时“溶解”;
而未照光或光酸耗尽的对照组中,纤维完好无损。
研究者相当于实现了一个“光控分子橡皮擦”:
通过紫外光的照射,就能在纳米尺度上精准地擦除、重构这些蛋白丝的分布。
这是一个非常未来感的概念——
用光操控分子结构,实现空间可编程的纳米装配。
4. 意义总结:从“响应”到“可控响应”
这一系列实验揭示出三个关键科学突破:
时间分辨率:首次在液相中实时捕捉到蛋白丝逐步解体的全过程;
灵敏度极高:pH 仅改变 0.1–0.3 即可触发显著动力学变化;
外部可控性:通过光酸实现局部、可逆、空间精准的响应。
这意味着,Baker 团队设计的体系不仅会“响应环境”,还可以被“人类控制”。
从此,“蛋白质材料”的设计不再只是被动的结构模仿,而开始走向主动的分子编程(programmable molecular engineering)。
五、总结与展望
这项工作展示了 de novo 蛋白设计 在构建环境响应性材料方面的最新进展。
研究团队通过理性设计,实现了一个能够在极窄 pH 范围内可逆组装的螺旋蛋白丝体系,并在原子层级上验证了设计与实验的一致性。
该体系的几个关键特征值得强调:
结构精度高 —— cryo-EM 结构与设计模型几乎完全一致(界面 RMSD 仅 1.4 Å);
响应极为灵敏 —— 仅 0.3 个 pH 单位的变化即可触发丝的快速解装(<1 s);
可逆且可控 —— 通过改变组氨酸数量或引入光酸,可实现对响应点与空间分布的精确调控。
这些成果说明,蛋白设计已从“稳定结构的构建”迈向“动态功能的控制”。
通过结合计算设计与实验验证,研究者实现了对自组装蛋白体系响应行为的定量可编程化。
未来,这类体系有潜力应用于:
智能药物递送与可控释放,利用 pH 或光信号实现空间与时间选择性;
可逆水凝胶或生物复合材料,在组织工程与再生医学中提供环境敏感的结构支撑;
纳米级信号响应组件,作为合成生物系统或分子机器人中的动态模块。
总体来看,这篇工作为“可预测、可调控的蛋白自组装材料”提供了清晰的设计路线,也标志着计算蛋白设计正从结构可控进入功能可控的新阶段。
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →