ICML 2025|UniMoMo:多肽、小分子与抗体设计的统一生成框架
蓝极说:
今天要分享的是一篇今年ICML 2025的关于多肽、小分子、抗体设计的工作,来自清华大学与字节跳动合作团队:
《UniMoMo: Unified Generative Modeling of 3D Molecules for De Novo Binder Design》,第一作者为 Xiangzhe Kong(清华大学)。
这篇论文提出了一个可以同时设计 小分子、肽和抗体结合体 的统一生成框架——UniMoMo,在多个任务上超越现有专用模型,被认为是跨分子类型生成的一次重要突破。
一、一个模型,设计所有分子?——UniMoMo的野心
在分子设计领域,不同类型的药物通常意味着完全不同的模型体系。
设计小分子药物,需要关注官能团的组合与几何约束;
设计肽药物,要考虑链式序列的折叠;
而抗体设计,又是一整套全新的结构逻辑与能量规则。
于是我们习惯性地认为——小分子、肽、抗体是三条平行的“AI 赛道”。每个赛道都有自己的生成模型、数据集和评估体系。
但如果我们能打破这三道墙呢?
这正是UniMoMo这篇文章中提出的设想:
能否用一个统一的生成框架,设计出不同类型的结合分子?
它能在给定同一个蛋白靶点时,同时生成肽、抗体、小分子三种类型的结合体。
在图 1 中,作者清晰地展示了 UniMoMo 的核心能力:
同一个靶蛋白的结合口袋(Target Protein),可以分别对接小分子、肽和抗体。
传统上,这三种分子的生成模型是彼此独立的;而在 UniMoMo 中,它们由同一个生成模型统一完成。
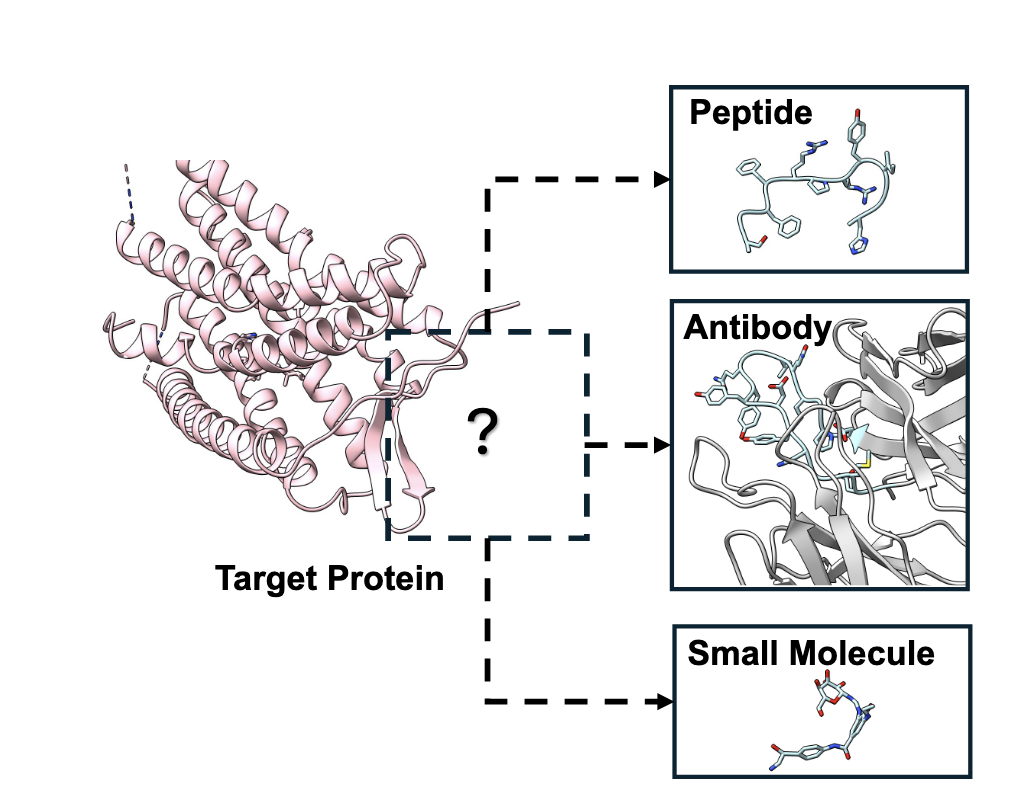
这种“多模态绑定生成”(multi-domain binder generation)不仅意味着技术整合,更暗示着药物发现范式的转变:
从“为每类分子造模型”,到“一个模型探索所有分子空间”。
二、为什么“统一”如此困难?
乍一看,“一个模型生成所有类型的分子”听起来只是把任务并到一起。但对分子设计来说,这几乎是一个跨物种级别的挑战。
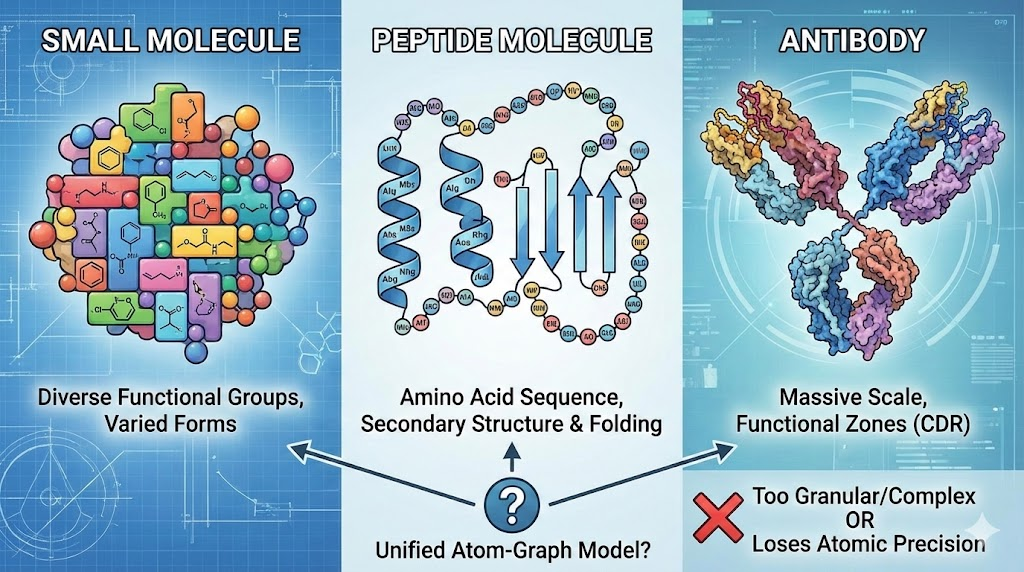
三类分子,三种世界
每种分子类型都有自己的语言和逻辑:
小分子:由多种官能团拼接而成,形态千变万化;
肽分子:由氨基酸顺序线性排列而成,更强调二级结构与折叠规律;
抗体:不仅包含氨基酸主链,还分布着功能分区(如 CDR 区域),结构规模庞大。
如果用同一种表示方法(例如“原子图”)去建模它们,就会出现两个极端问题:
要么过于细碎、计算复杂;要么失去原子层级的几何精度。
图片生成于Nano Banana
简单来说,小分子和抗体的“构造方式”完全不在一个维度上。
想用一套生成模型同时处理这两种结构,几乎就像让语言模型同时写英语诗和化学方程式。
⚙️ 挑战背后:层级结构与几何约束的冲突
分子结构遵循一套通用的物理化学规则——键长、键角、空间排斥、氢键方向性……这些规律跨越分子类型而存在。
但它们在不同的结构层级上表现得截然不同。
小分子里,规则约束在局部官能团上;
抗体中,这些约束分布在成百上千个原子组成的模块内。
所以,真正的挑战是:如何在同一个框架下既保留原子级细节,又捕捉分子间的层次结构。
从“原子”到“块”:UniMoMo 的核心思想
作者的关键洞察是:
“不同类型的分子,其实都可以拆解为若干个可复用的结构单元(blocks)。”
于是,UniMoMo 引入了一个全新的分子表示方式——Graph of Blocks。
每个分子被拆分为多个“块”:
如果是肽或抗体,每个“块”就是一个标准氨基酸;
如果是小分子,则用算法自动提取常见的官能团或环状结构,如苯环、吲哚等。
然后将这些“块”拼接成一个统一的图结构。
这样的设计同时满足了两种需求:
在宏观层面保留了结构的层级性;
在微观层面保留了每个块的原子细节。
这正是 UniMoMo 统一建模的基础,也为后文的几何扩散生成(Geometric Latent Diffusion)提供了表示支撑。
三、从“原子云”到“积木图”:UniMoMo 的统一语言
在前一节,我们讲到不同分子类型有着各自的“结构语言”。小分子用官能团在说话,肽和抗体则用氨基酸在表达。
如果 AI 想同时理解这三种语言,第一步就得找到一种通用的表达方式。
这正是 UniMoMo 的第一项关键创新——
用“Graph of Blocks”重新定义分子表示。
一种跨分子通用的表示法
论文中的 Figure 2A 给出了这个思想的视觉化解释:小分子、肽、抗体——都被拆分成一个个“Block”。
每一个 Block 代表一个局部结构单元,可以是:
一个标准氨基酸(对肽或抗体);
一个常见分子片段,如苯环、吲哚环、酰胺基(对小分子)。
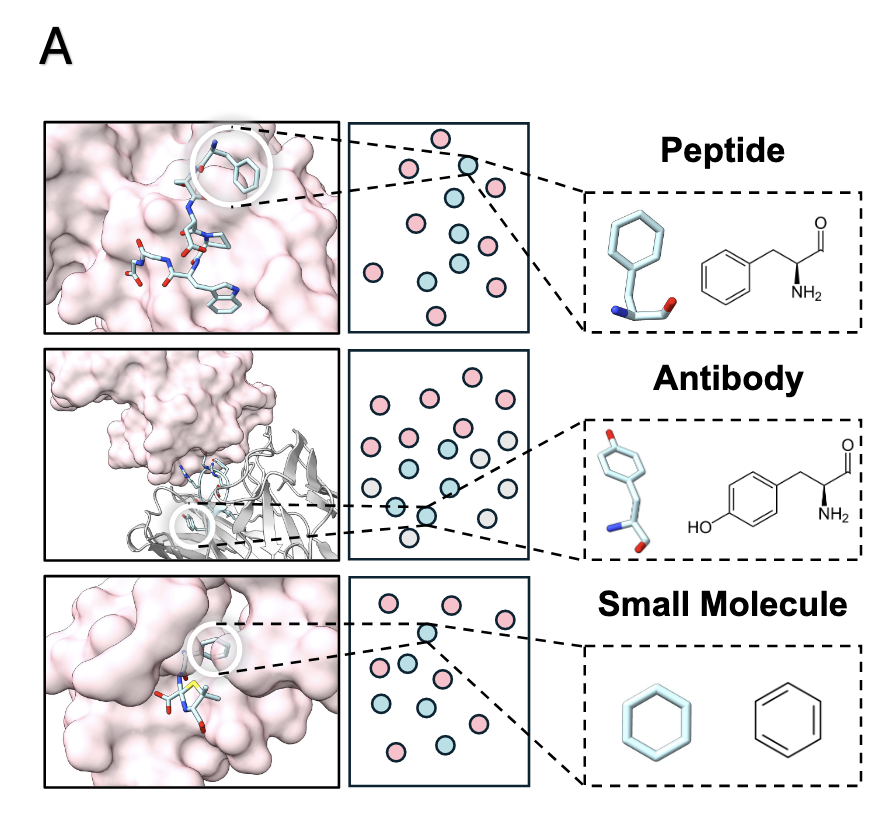
这些 Block 再通过化学键(bond)相互连接,
于是无论是复杂的抗体,还是微小的小分子,都能被统一地表示为一个由 Block 组成的图(Graph)。
简而言之:肽是由氨基酸 Block 拼起来的“小分子”,小分子是由官能团 Block 拼起来的“肽”。
关键算法:Principal Subgraph 分解
那问题来了——AI 怎么知道哪些部分可以当作“Block”?
作者在2022年的NeurIPS提出了一个自动化的结构分解算法:
Principal Subgraph Mining(主子图提取)。
它会在海量分子结构中寻找出现频率最高的局部结构(例如苯环、咪唑环、酰胺键),并将它们定义为标准化 Block 词汇。

最终,模型拥有一个“分子词典(Vocabulary)”,里面既包括 20 种氨基酸,也包含常见的化学基团。
每个分子被分解后,AI 就能用统一的“Block 词汇表”来描述所有类型的分子。
这一步的意义,就像自然语言处理中的“分词”:
它把连续的原子结构,切分成有化学意义的片段单元。

表示优势:兼顾层次与精度
这种 Block Graph 表示 带来了两个决定性的优势:
- 保留原子精度:
每个 Block 内部仍然存有完整的原子坐标、键长、键角等信息,因此模型可以在后续阶段恢复出全原子级几何结构。
- 降低复杂度,提升泛化:
对抗体这样的庞大体系,直接处理原子图几乎不可能;而用 Block 级表示后,模型只需学习模块间的组合规律,就能在不同分子类型之间迁移学习。
四、从“潜空间粒子”到“立体分子”:UniMoMo 的生成之心
当我们用 Block Graph 统一了分子语言后,下一步的问题是——AI 如何“说”出新的分子?
在 UniMoMo 中,这个“说话”的过程被分为两步完成:
先把所有 Block 压缩成潜空间粒子(latent points);
再通过一个几何扩散过程(geometric diffusion),在三维空间中生成并还原出完整分子。
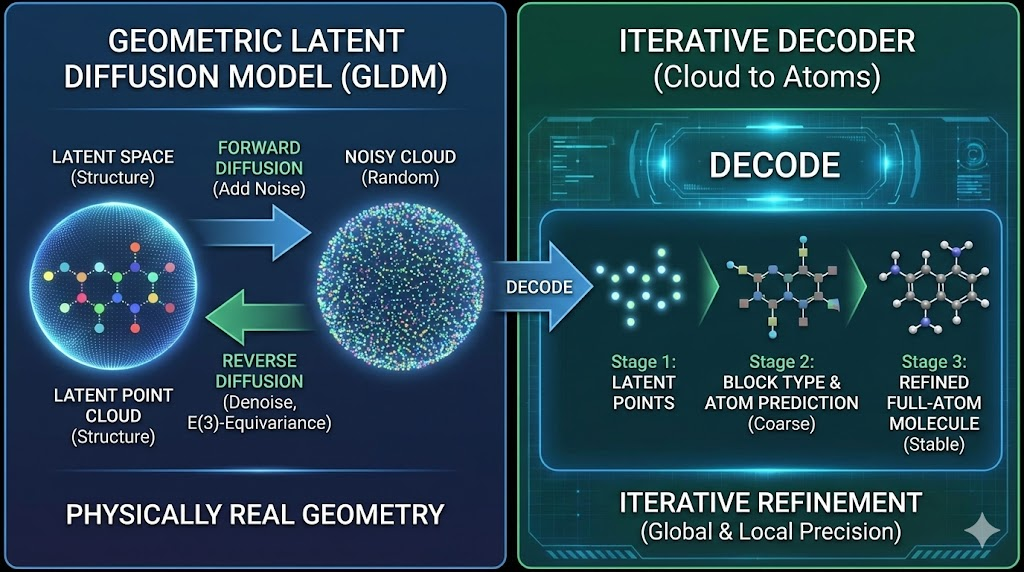
这一整套机制在论文的 Figure 2B 中有非常直观的图示——
左边是编码压缩(Encoder),中间是扩散生成(Diffusion),右边是逐步还原结构(Decoder)。

第一步:AutoEncoder 压缩——让分子“隐入潜空间”
在传统分子生成模型中,AI 直接操作原子坐标,这不仅计算量庞大,还难以处理复杂体系(尤其是抗体)。
UniMoMo 采用了一个名为 Iterative Full-Atom Variational AutoEncoder 的机制。
这个模块的任务是:
把每个 Block 压缩成一个带空间坐标的小点(latent point)。
具体来说:
Encoder 将 Block 的原子信息(类型 + 坐标)映射到一个隐向量 $$z_{i}$$和一个三维位置 $$\vec{z}_i$$;
这些隐点构成了一个“潜空间点云(latent point cloud)”,
它的几何分布依然反映了原始分子的空间结构。

图片来源:Nano Banana
可以把这一步想象成:
“每个分子被压缩成一团粒子云,每个粒子代表一个局部结构单元。”

第二步:扩散生成——在潜空间中“呼吸”出新分子
一旦模型把真实分子压缩到了潜空间,就能在这个空间里学会生成新的点云分布。
这一步使用的是一种 几何潜空间扩散模型(Geometric Latent Diffusion Model, GLDM)。
它的工作原理类似于“呼吸”:
正向扩散(Forward Diffusion):往潜空间点云中逐步加入噪声,直到一切变成随机云团;
反向扩散(Reverse Diffusion):AI 学会一步步去噪,还原出符合化学约束的点云结构。
与传统的扩散模型不同,UniMoMo 的扩散过程具有 E(3)-等变性(E(3)-Equivariance):
也就是说,无论整个分子在三维空间中如何旋转或平移,
模型生成结果的几何关系都不会被破坏。
这点非常关键——它保证了生成的分子在几何上是“物理真实”的,而不是数学伪像。

第三步:Decoder 还原——从点云到全原子结构
当潜空间中的点云被扩散模型生成完毕,模型就需要重新“展开”成原子的层级。
UniMoMo 的 Decoder 是一个迭代重建器(Iterative Decoder):
先根据潜点的隐向量预测每个 Block 的类型(如苯环、亮氨酸等);
再一步步预测每个原子的坐标与化学键。
这个过程并非一次性完成,而是多轮递进式的“细化”:
每一轮都修正坐标、预测化学键、优化几何关系,直到生成一个稳定的三维分子。
这让模型能够在保留全局构型的同时,兼顾原子级局部精度。
就像艺术家先画出骨架,再逐层上色,最后雕刻出化学细节。
图片来源:Nano banana
为什么这套机制高效又强大?
这套“AutoEncoder + Diffusion”的设计有两个显著优点:
- 降维带来高效
Diffusion 不再在原子层级运行,而是在压缩后的潜空间进行,大大降低了计算量,让抗体级别的分子生成成为可能。
- 局部精细,整体合理
Decoder 负责细化每个 Block 的原子结构,而 Diffusion 专注于全局布局,二者分工明确,互相补强。
五、一个模型打三场仗:UniMoMo 的全领域表现
UniMoMo 不止是“能同时生成三类分子”,更关键的是——它在每一类任务上都更强。
作者在三条典型任务上进行了系统评测:
肽设计、抗体设计、小分子设计。
结果显示,这个统一模型不仅性能全面超越专用模型,还展现出跨领域迁移带来的几何与能量优势。
实验一:肽设计(Peptide Design)
在肽任务上,UniMoMo 被测试于多个 benchmark(PepBench、ProtFrag、LNR 等)。
研究者关注几个关键指标:
C-RMSD / L-RMSD:生成肽结构与真实肽的偏差;
ΔG(自由能)与 IMP(能量优越率):生成肽与蛋白结合的能量;
Clash rate 与 JSD:几何冲突率与二面角分布一致性。
在表格 Table 1 中可以看到:

UniMoMo 的 binding energy(-34.35 kcal/mol) 远低于所有对照模型(如 PepFlow、PepGLAD);
同时结构 RMSD 下降到 仅 2.19 Å,意味着它能生成更贴近天然肽构象的结合体;
更有趣的是,模型的 Clashout(肽与蛋白原子冲突率)低至 0.45%,比以往模型减少了数倍。
换句话说,UniMoMo 生成的不只是“像样”的肽,而是真正能稳稳贴合靶点表面的肽。
实验二:抗体设计(Antibody Design)
抗体的挑战在于 CDR(互补决定区)结构的复杂性。
传统模型往往只能生成 backbone 框架,而无法稳定预测侧链几何。
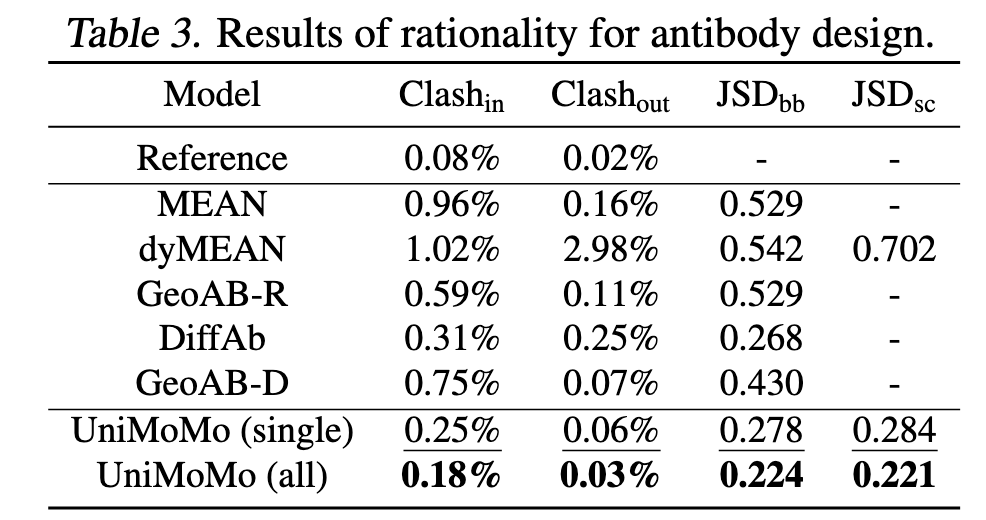
在 Table 2–3 中,UniMoMo 的表现非常突出:
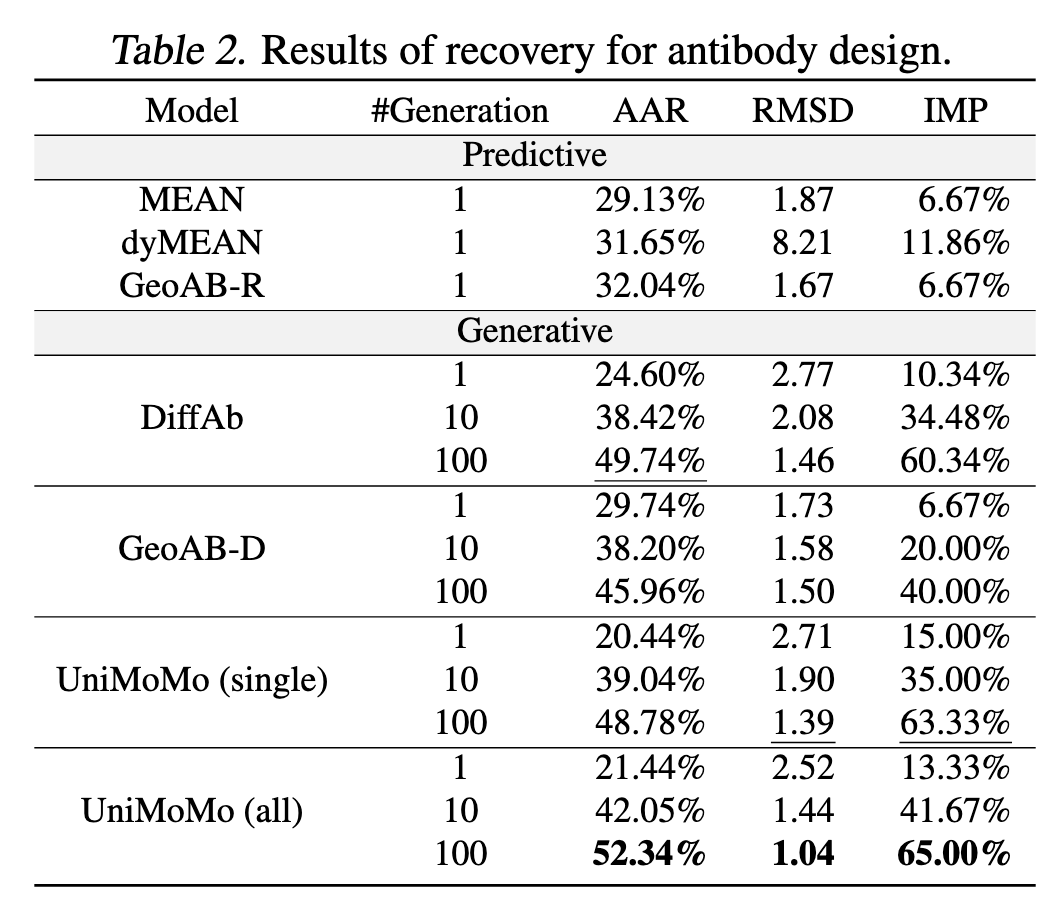
当生成 100 个候选抗体时,AAR(氨基酸恢复率)达到 52.3%,远超 DiffAb(49.7%)和 GeoAB(45.9%);
其 RMSD(结构偏差)仅 1.04 Å,已接近实验结构精度;
更关键的是,65% 的生成抗体在结合能上优于原始天然抗体。
几何质量同样令人印象深刻:
UniMoMo 的 Clashin / Clashout 比例极低(0.18% / 0.03%),说明生成的结构在原子层面几乎没有无效碰撞。
这意味着模型不仅能“生成抗体”,还真的在学习“免疫几何”。
⚗️ 实验三:小分子设计(Small Molecule Design)
小分子设计的评测最全面。
作者采用了 CBGBench 框架,从四个维度评估模型:
Substructure(结构单元一致性)
Chemical property(药物理化性质)
Geometry(几何合理性)
Interaction(蛋白结合模式)
在 Table 4 中,UniMoMo 拿下了总分第一名。
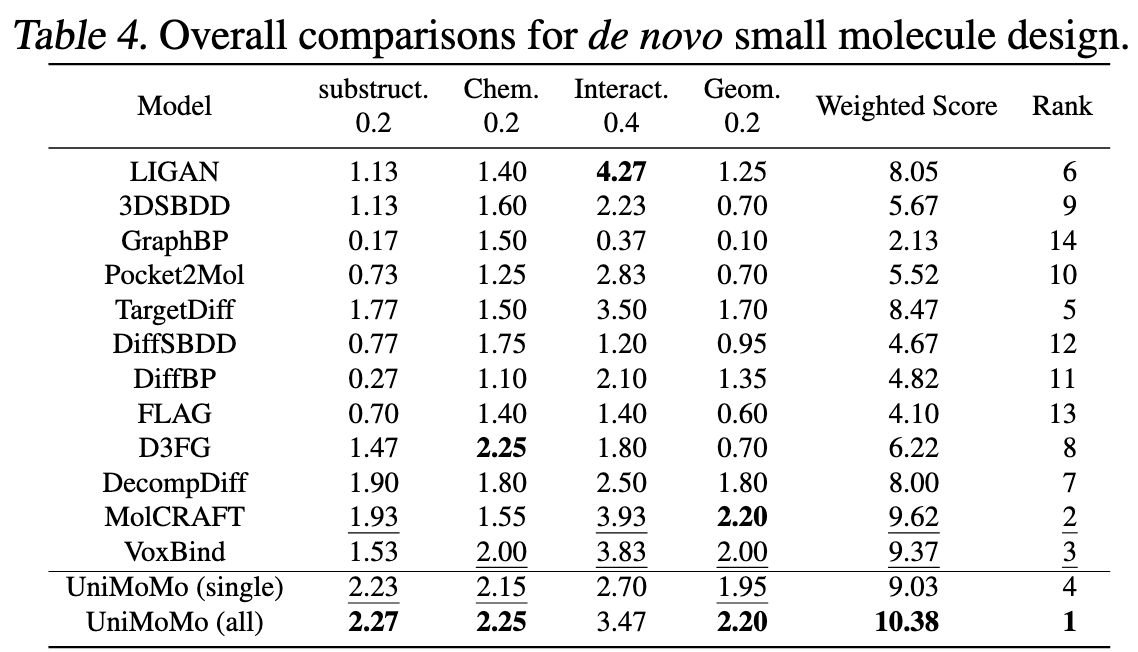
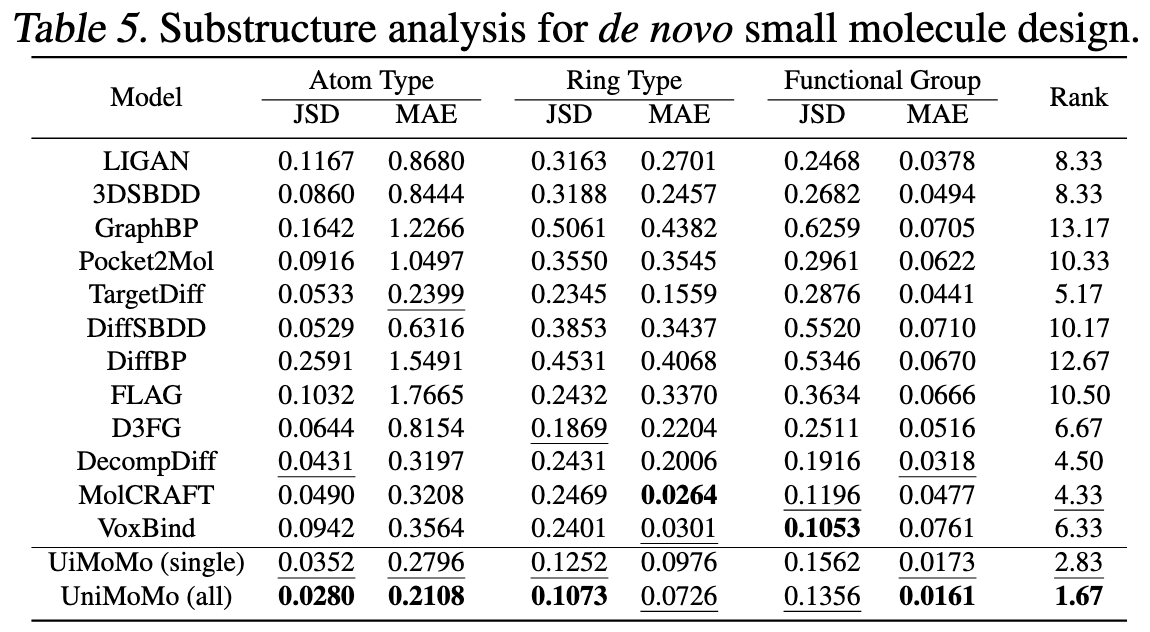
具体亮点包括:
- 在“结构子单元一致性”上(Table 5),UniMoMo 的 JSD 仅 0.028,说明它生成的原子与环结构分布几乎完美匹配真实分子;
- 在“药物可合成性”指标(SA 值)上,它也超越所有模型;
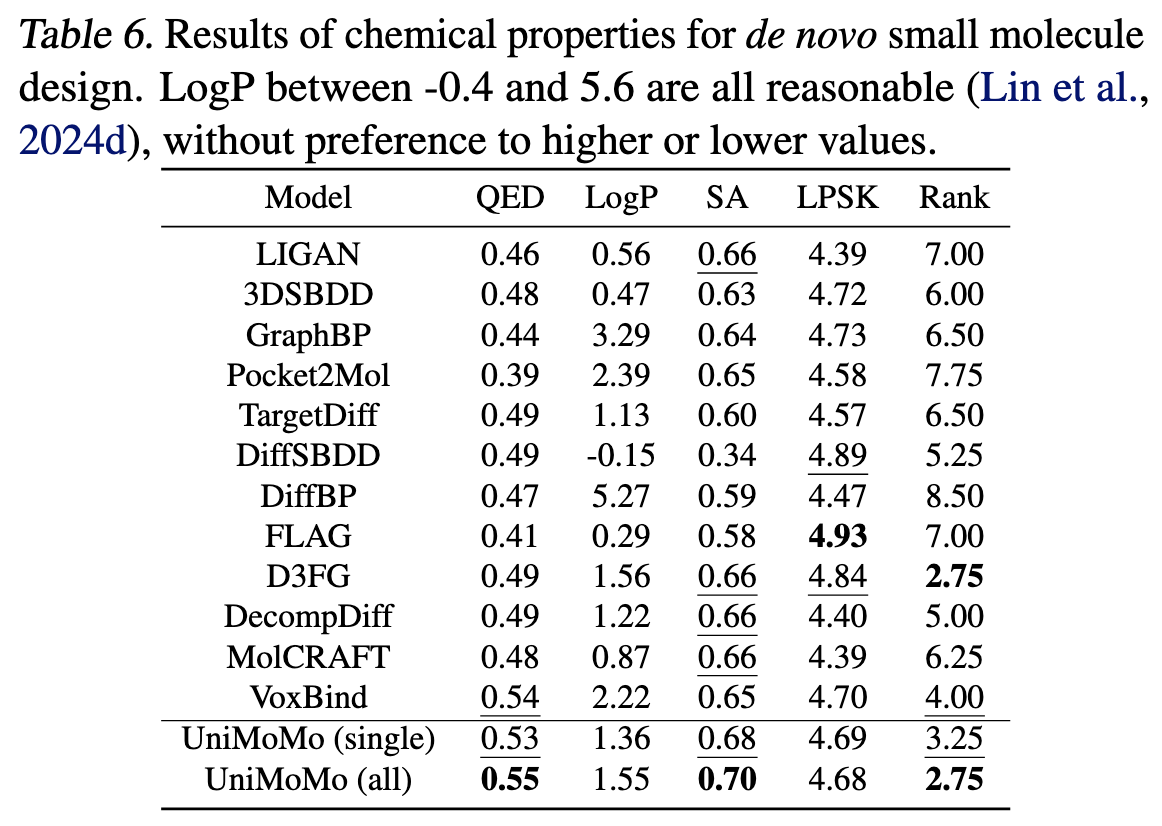
- 在“几何合理性”部分(Table 7),生成分子几乎无原子重叠(Clash ratio 仅 0.004)。
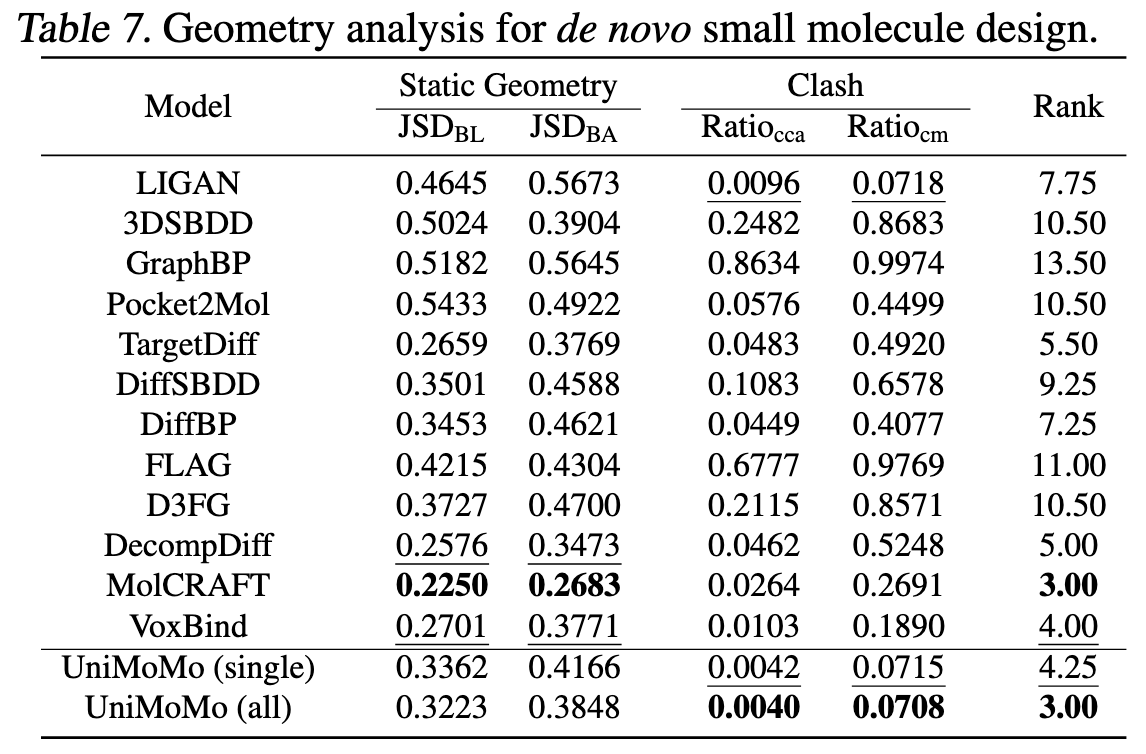
在这一领域,UniMoMo 不只是一个“通才”,而是一个能打的“全能冠军”。
Figure 3:同一个靶点,三种分子,三种策略
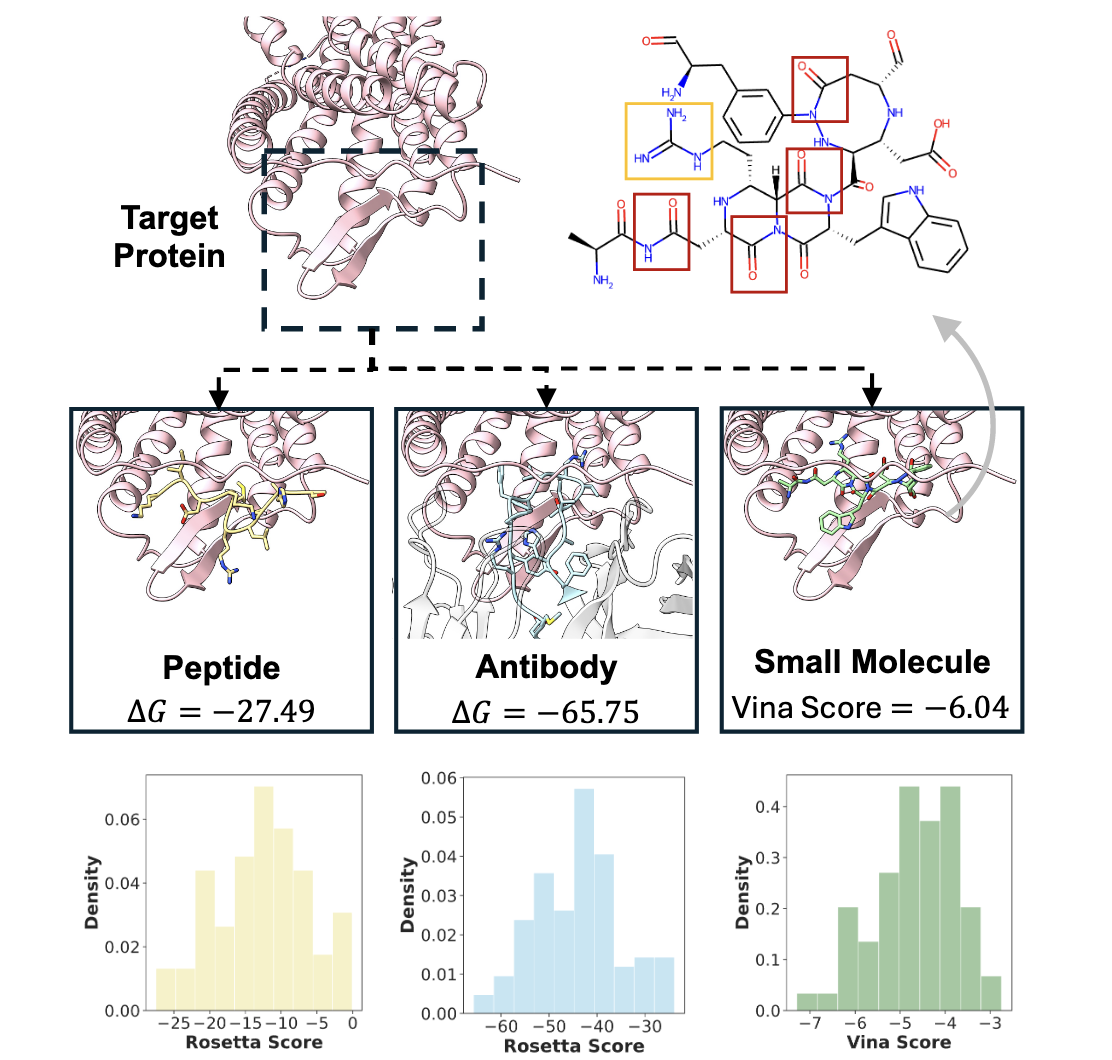
为了展示模型的“跨域迁移”能力,作者选取了一个GPCR(G 蛋白偶联受体),PDB ID: 8U4R。
UniMoMo 在同一个结合口袋上分别生成了三类分子:
肽:自由能 ΔG = -27.49 kcal/mol;
抗体:ΔG = -65.75 kcal/mol;
小分子:Vina 打分 = -6.04 kcal/mol。
更有意思的是,论文指出两个令人惊喜的观察:
小分子中出现了模仿氨基酸侧链(如精氨酸)的结构;
生成的骨架还出现了类似抗体肽链的酰胺连接片段。
换句话说,模型在“不同分子类型之间学会了借鉴”。
它能从抗体中学到空间布局,再把这种“几何知识”迁移到小分子设计中。
这正是 UniMoMo 真正“统一生成”的核心意义。
小结
通过三类任务的系统对比,UniMoMo 展示出一个清晰的趋势:
“统一建模”并不是在三种任务之间妥协,而是在三种任务之间形成协同。
多域数据训练让模型学到更通用的分子几何规律,进而反哺到每一个子任务的生成质量。
六、从多域生成到通用分子智能
当我们重新回看这篇论文,会发现它的意义远不止“性能更强”。
UniMoMo 的真正突破在于,它第一次证明了:
分子设计的通用生成模型是可行的。
过去我们习惯让模型在各自的“生态位”内训练——
小分子学有机化学,肽模型学蛋白折叠,抗体模型学免疫几何。
而 UniMoMo 则在同一个潜空间中,让这三种分子共享几何知识、互相迁移结构经验。
这种“跨域共学”的设计,不仅让模型在多种任务上表现更好,也暗示着一个更深的科学图景:
AI 可能正在学习分子世界的共同语法。
UniMoMo 给我们提供的不仅是一个算法框架,更是一种新的思维方式:
让模型自己去发现分子世界的共性。
当 AI 具备这样的能力,它将不只是加速药物研发,而是改变我们理解生物体系的方式。
原文链接:https://openreview.net/pdf?id=KUN7A7Okb6
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →