Click Fusion:基于人工设计融合蛋白的 GPCR 非活化态结构解析新策略
今天介绍的这篇文章非常有意思,是我认识的一个师兄的文章。文章非常有新意,在GPCR蛋白质设计和结构生物学这个领域另辟蹊径,从蛋白质设计到结构解析再到蛋白质工程几个领域都做了很不错的尝试,让我们一起来读一读吧!

原文链接:https://doi.org/10.1073/pnas.2422360122
1. 研究背景
G 蛋白偶联受体(G protein–coupled receptors, GPCRs)是一类典型的七跨膜结构膜蛋白,能够识别包括激素、神经递质、光信号等在内的多种外界刺激,并将信号传递到细胞内部,在生理调控中发挥核心作用。由于其在疾病发生发展过程中的重要性,GPCR 也是药物研发的重点靶点。据统计,约 34% 的 FDA 批准药物以 GPCR 为作用靶点,其中拮抗剂类药物占比超过一半。
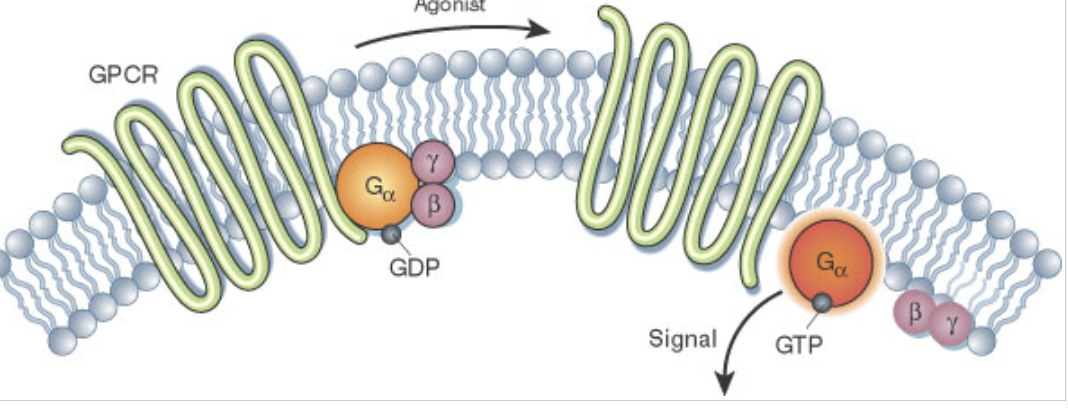
GPCR 拮抗剂通过稳定受体的非活化构象(inactive state)来阻断信号转导,因此解析 GPCR 在非活化状态下的高分辨率结构,对于阐明作用机制和指导结构基础上的药物设计具有重要意义。然而,获取 inactive 状态 GPCR 结构仍面临技术难题。与激动剂结合的活化态结构不同,非活化态 GPCR 缺乏与 G 蛋白复合形成的稳定支架,分子量较小且整体柔性高,在单颗粒冷冻电镜(cryo-EM)数据处理中难以进行可靠的粒子对齐。此外,已有的融合蛋白或纳米抗体稳定策略往往需要大量的实验筛选和结构优化,工作量大且适用范围有限。
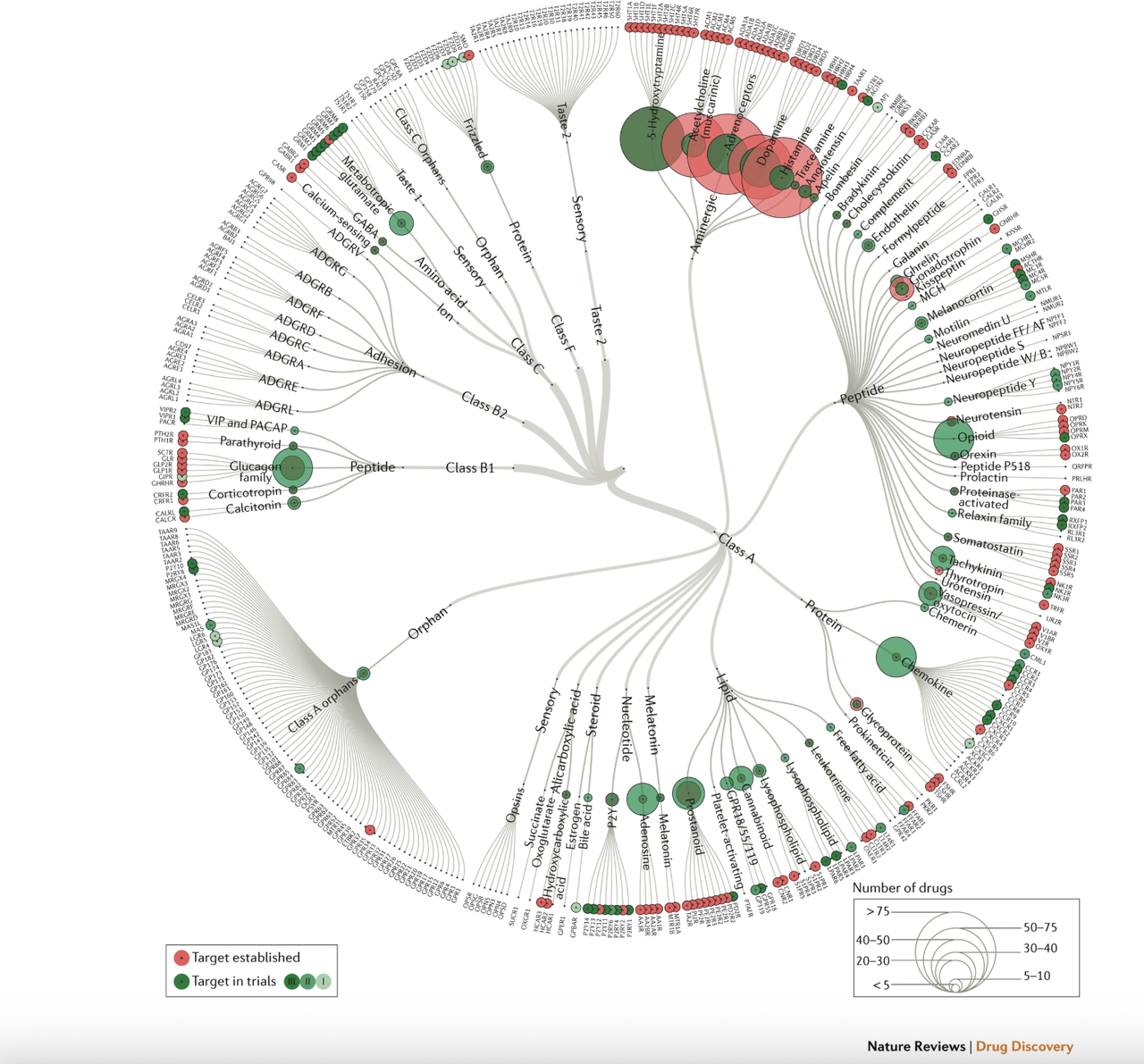
本研究针对这一难题,提出了一种基于人工设计融合蛋白的新策略——Click Fusion。该方法通过将一个刚性蛋白结构域以精准设计的连接方式固定在 GPCR 的 TM5 和 TM6 之间,从而提高整体稳定性,并为 cryo-EM 数据处理提供显著的结构特征信号。同时,这一融合蛋白具有可迁移性,可在结构类似的 GPCR 间直接应用,大幅降低构建和优化的工作量。
2. 创新方法:Click Fusion 设计策略
为解决 inactive 状态 GPCR 结构解析中支架不足的问题,作者提出了一种 de novo 蛋白设计 的融合策略——Click Fusion。该方法的核心是构建一个高刚性融合蛋白(Clip),精准连接于目标 GPCR 的跨膜螺旋 5(TM5)与跨膜螺旋 6(TM6)之间,以增强整体构象稳定性并提供 cryo-EM 对齐所需的可识别特征域。
设计流程如 Fig.1 所示,分为以下几个关键步骤:
融合位点选择
基于 GPCR 结构模型(来自 AlphaFold2 预测或 GPCRdb 数据库),删除细胞内环 3(ICL3),并确定其与 TM5/TM6 的连接残基作为融合位点。
该位置选择可兼顾对受体天然构象的最小干扰与融合蛋白刚性传递的最大化。
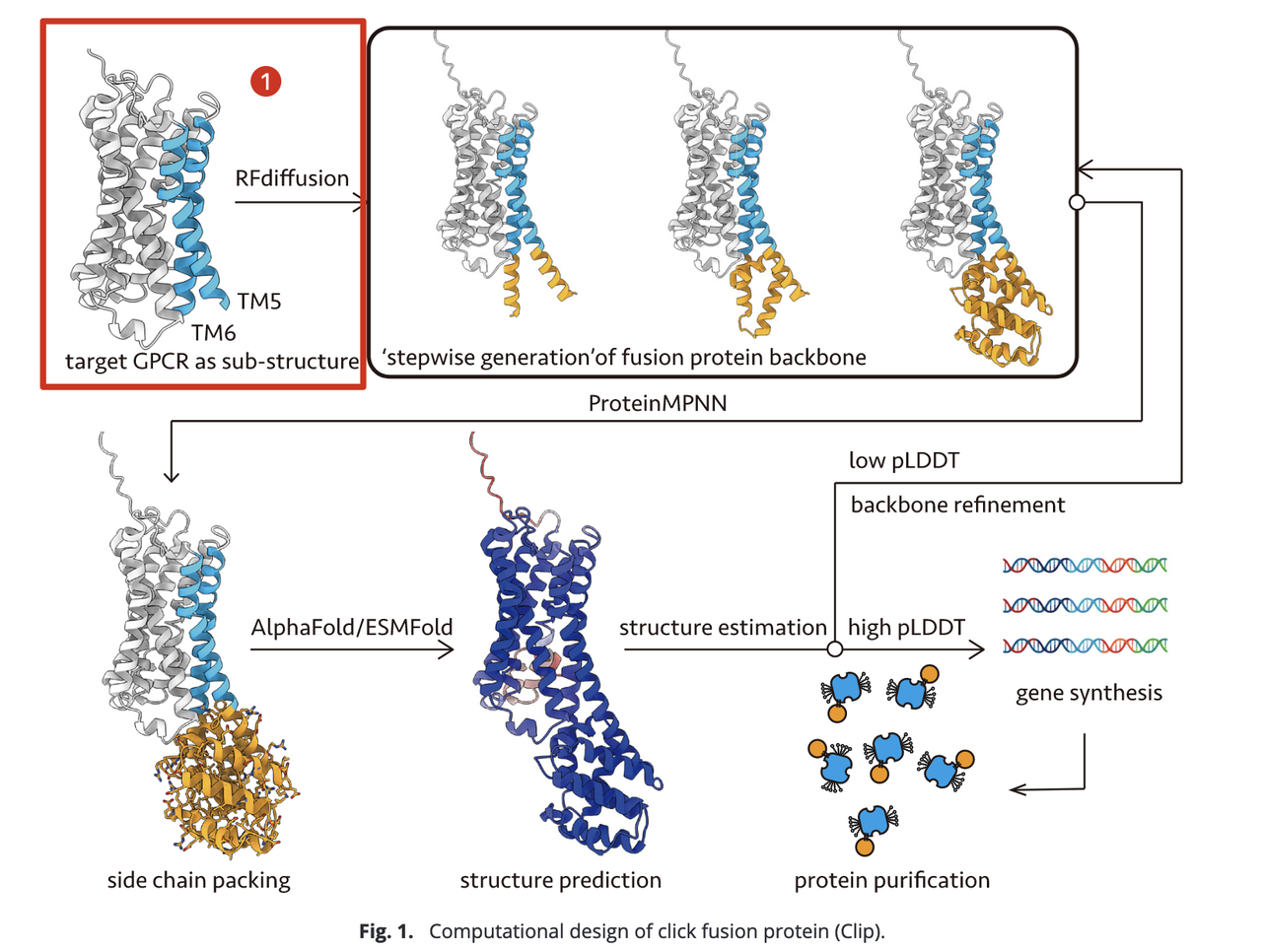
- 骨架生成(Backbone generation)
使用 RFdiffusion 方法分三步生成融合蛋白骨架:
延伸 TM5/TM6 的螺旋段;
在延伸区插入稳定中间螺旋,作为结构支撑;
生成完整的 Clip 蛋白骨架(长度约 100–150 aa)。
这种逐步构建方式可有效避免单步生成时融合域向膜内折叠的倾向。
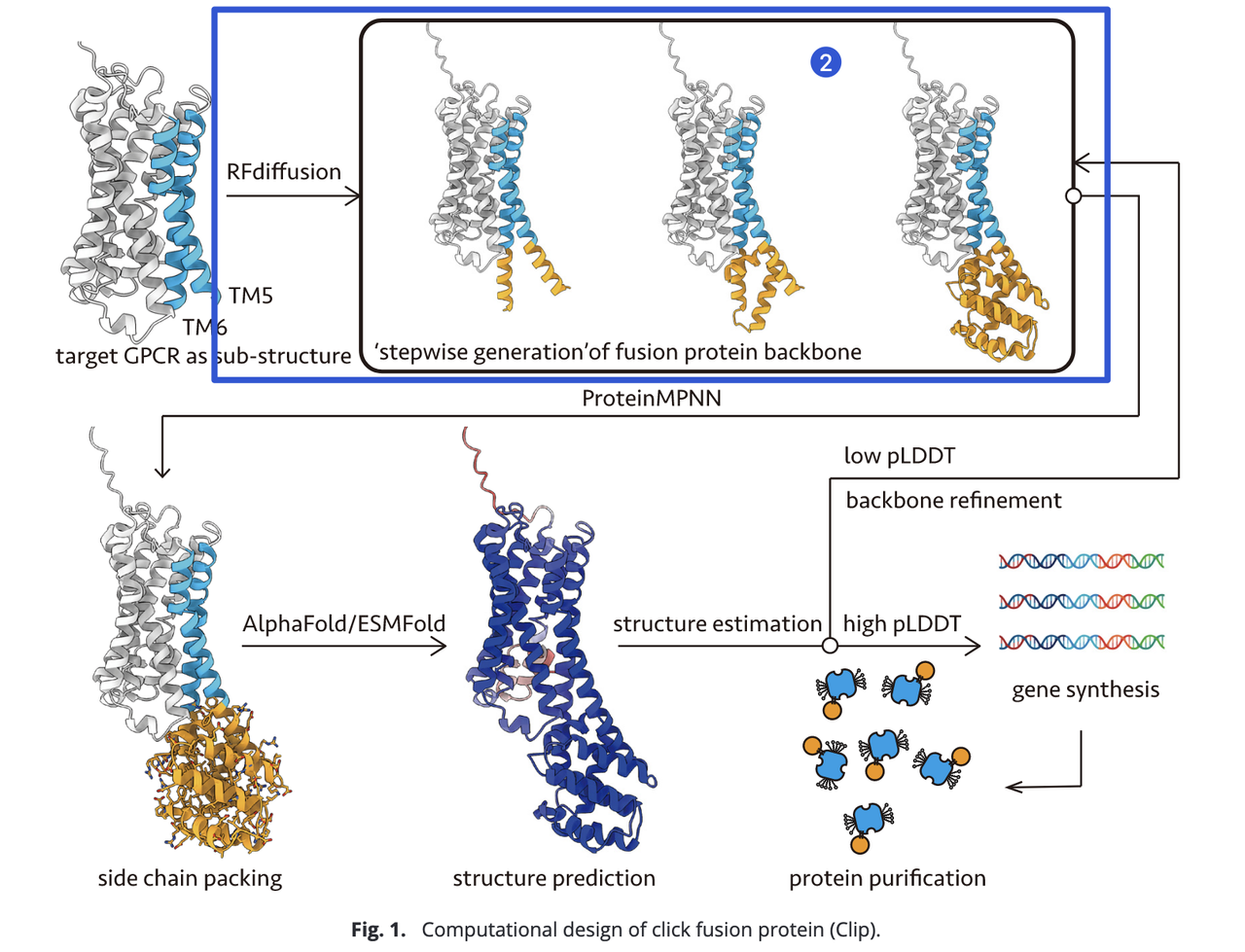
- 序列优化与结构预测
使用 ProteinMPNN 进行侧链打包与序列优化。
采用 AlphaFold2 或 ESMFold 对融合构建进行结构预测,并以 pLDDT 分数评估结构可信度和连接区域的刚性。
对低评分区域再次利用 RFdiffusion 优化骨架结构。
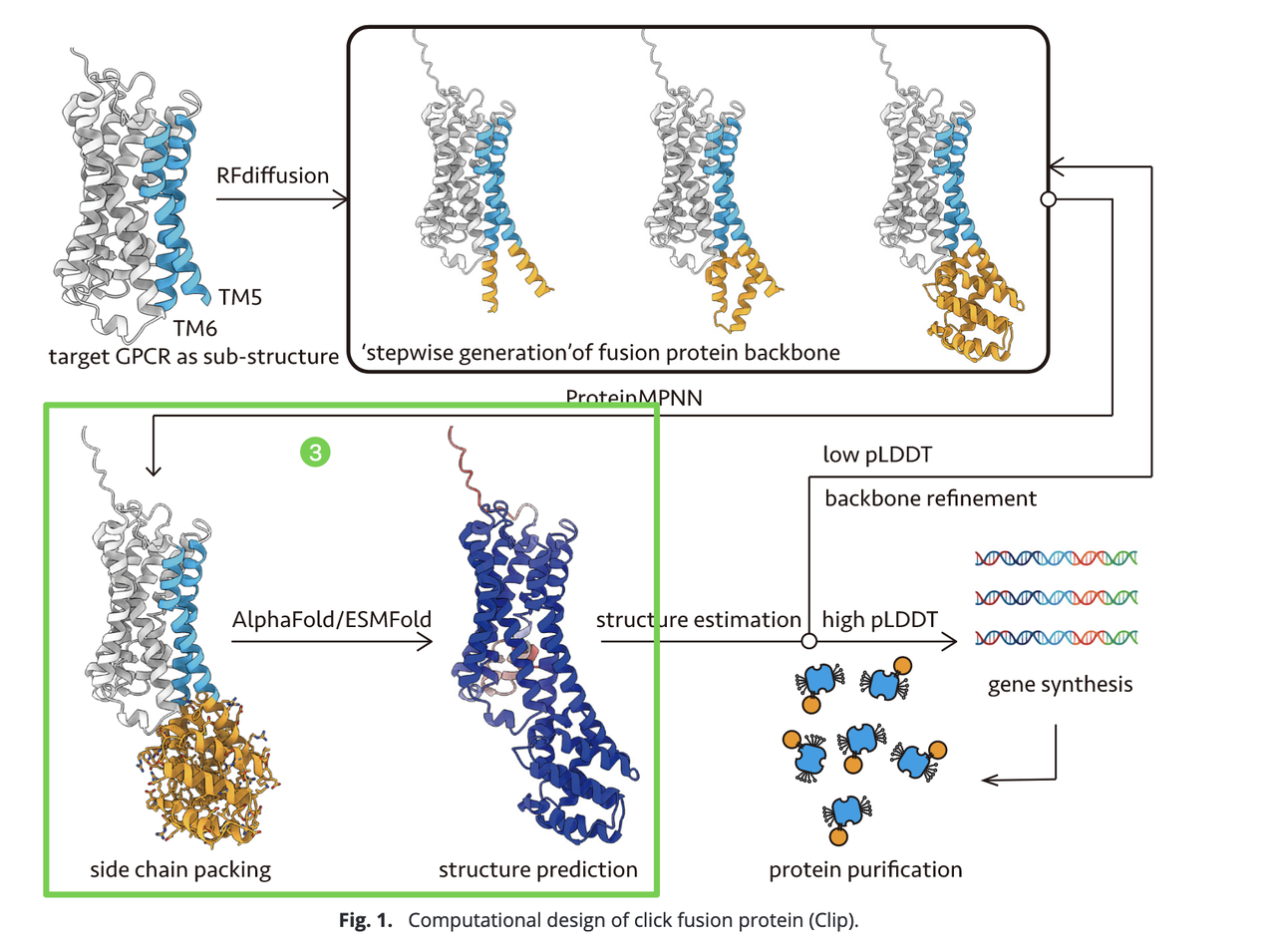
- 实验验证准备
- 选择高 pLDDT 分数的设计方案合成编码 DNA,并在细胞中表达、纯化以获得 GPCR-Clip 蛋白,进入后续生化与结构验证环节。
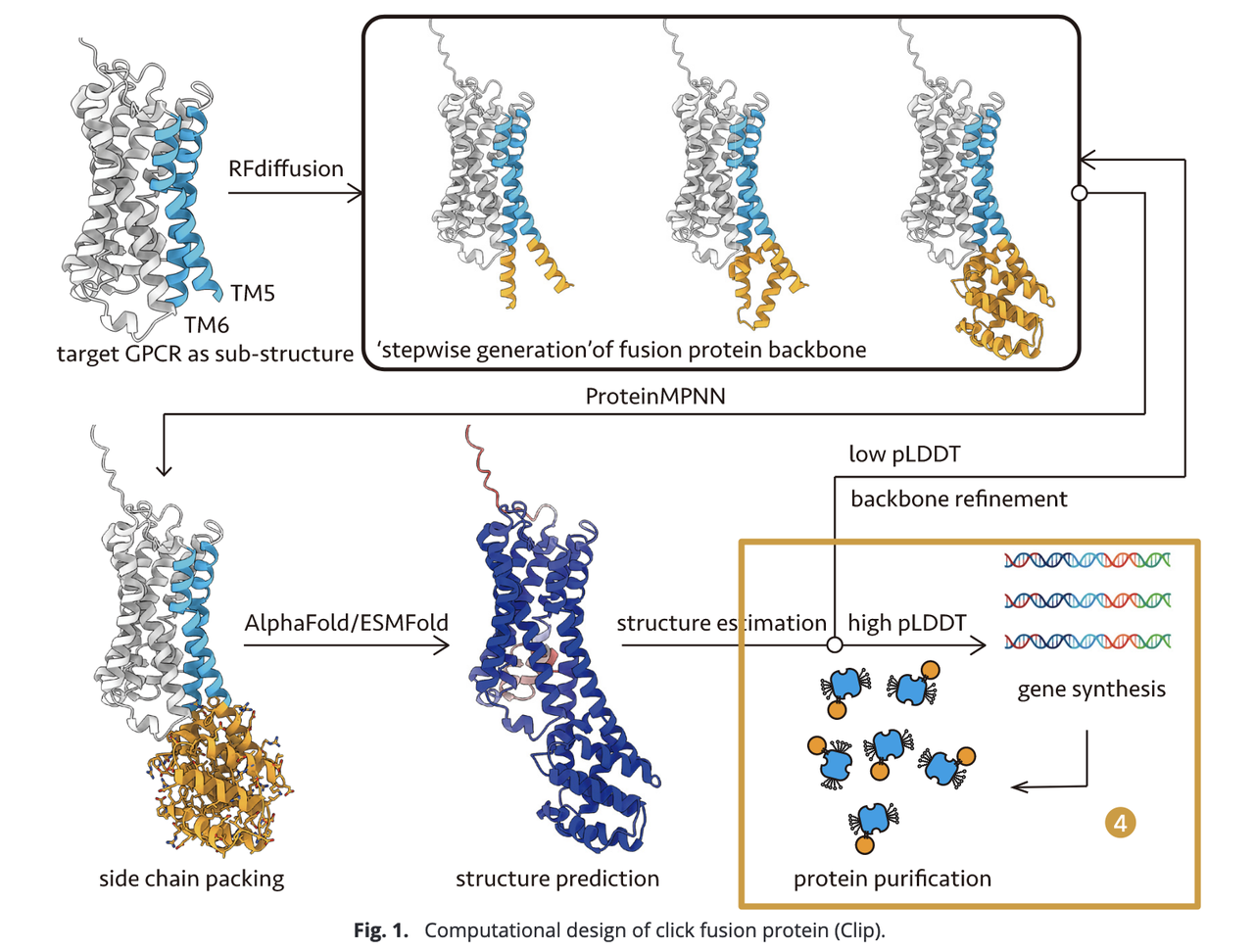
Click Fusion 的命名有两层含义:一是整个设计流程主要通过计算实现,相比传统方法大幅减少实验迭代,仅需“点击”鼠标即可完成大部分工程;二是 Clip 蛋白与 GPCR 的 TM5/TM6 区域在结构上高度匹配,像“卡扣”一样稳固结合。
该方法不仅适用于单一 GPCR 的定制化稳定,还可将设计完成的 Clip 蛋白直接迁移至结构相似的 GPCR,从而显著提升构建效率和通用性。
3. M1R 案例验证
为验证 Click Fusion 策略的可行性,作者首先选择了M1 型毒蕈碱乙酰胆碱受体(M1R)作为测试对象。M1R 属于典型的 class A GPCR,与学习、记忆和认知等多种神经功能相关,是阿尔茨海默病、精神分裂症以及药物成瘾等中枢神经系统疾病的重要潜在靶点。
- 表达与配体结合特性(Fig.2A、B、G、H)
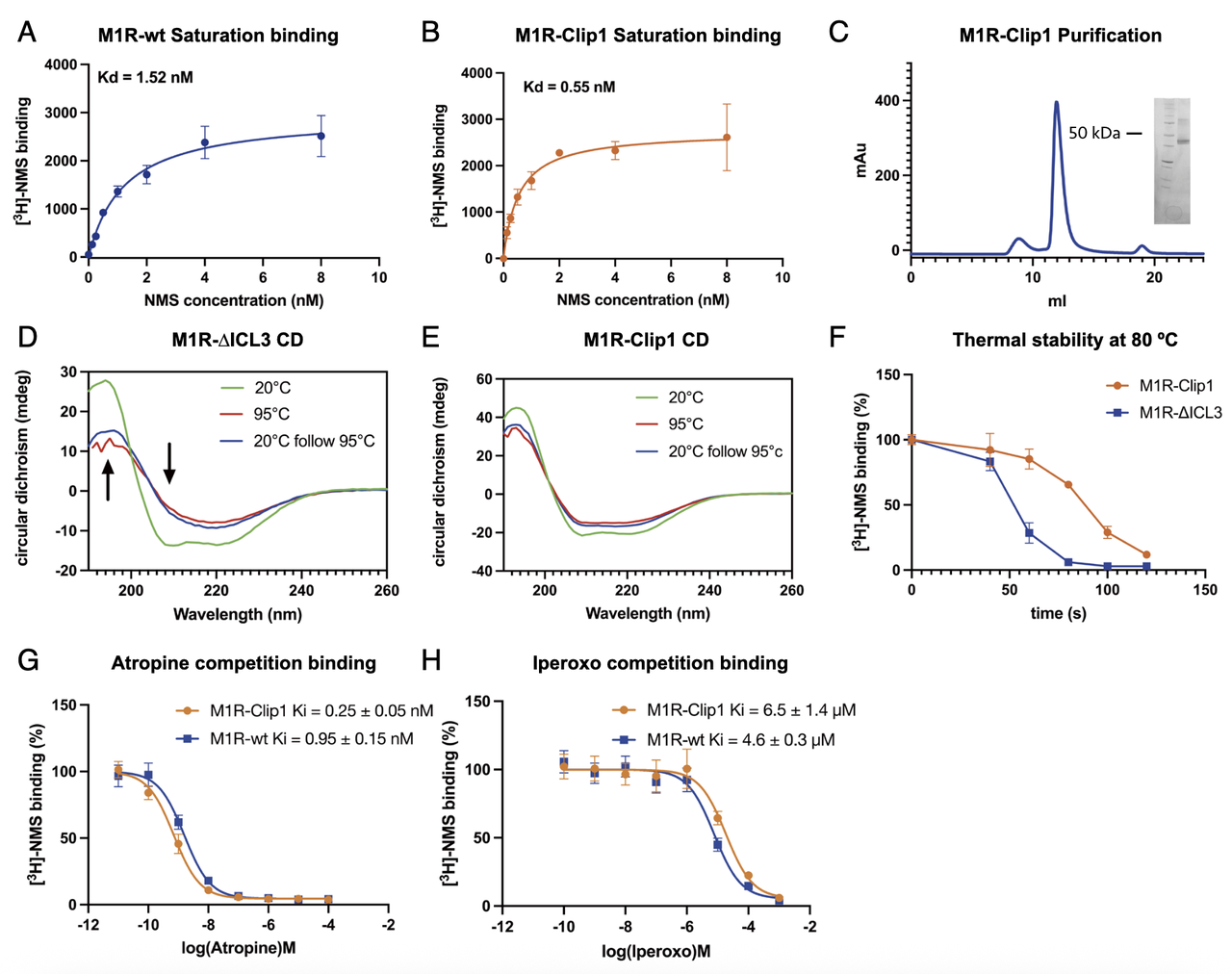
将 Clip1 融合于 M1R(M1R-Clip1 构建),并与已知高表达的 ICL3 缺失构建(M1R-ΔICL3)比较。
细胞表面标记实验表明,M1R-Clip1 的表达水平与 M1R-ΔICL3 接近,显著高于野生型 M1R。
放射性配体饱和结合实验([³H]-NMS)显示,M1R-Clip1 与野生型 M1R 的解离常数(Kd)相近(0.55 nM vs. 1.52 nM),说明融合不影响受体与拮抗剂的亲和力。
配体竞争结合实验中,M1R-Clip1 对代表性拮抗剂 atropine 和激动剂 iperoxo 的结合能力与野生型 M1R 基本一致,进一步证明其保留了原有药理特性。
热稳定性提升(Fig.2D、E、F)
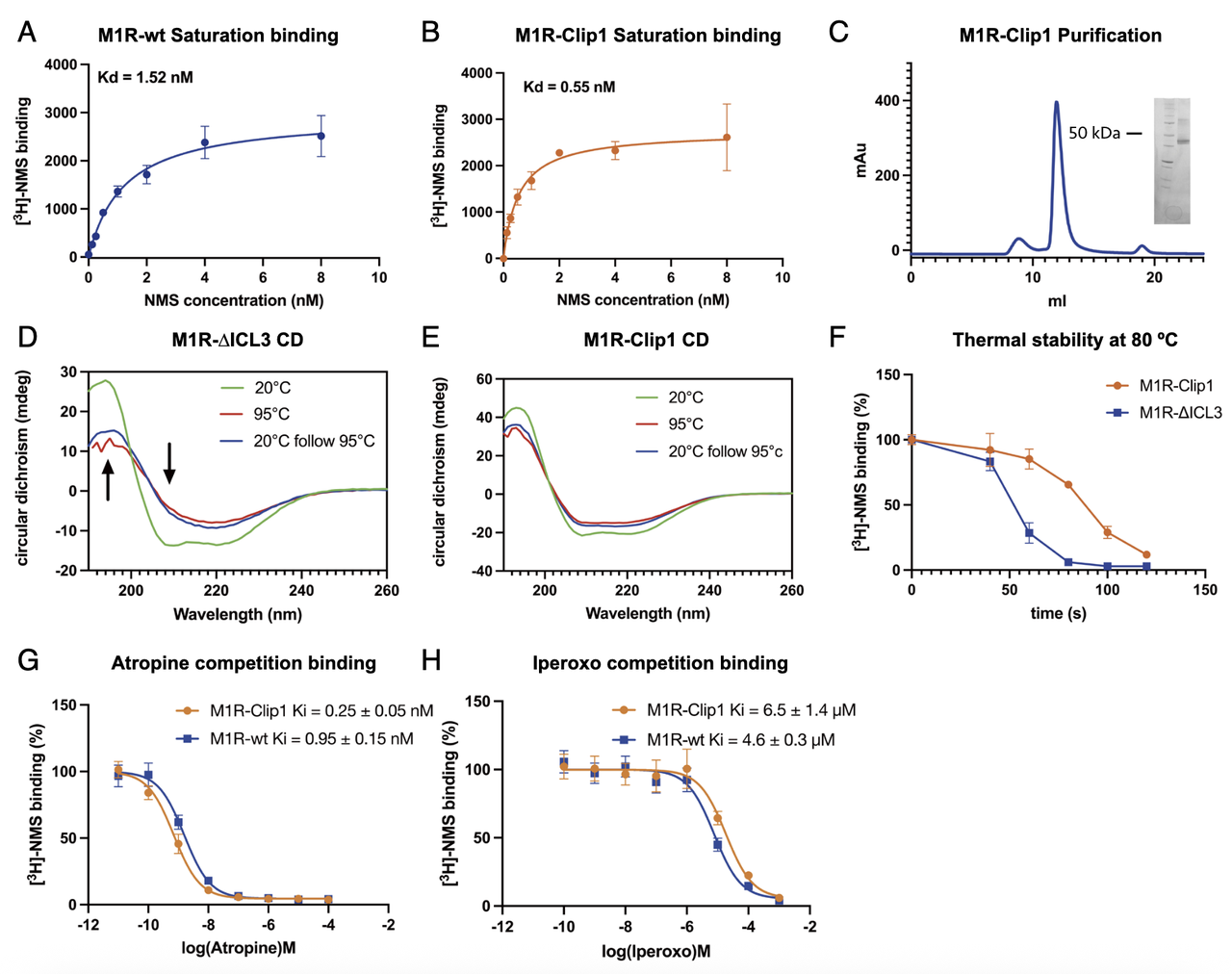
圆二色谱(CD)谱图显示,M1R-Clip1 在高温(95 °C)处理后仍能保持 α-螺旋特征,并在降温后恢复光谱信号;相比之下,M1R-ΔICL3 在加热后螺旋结构显著丢失且不可逆恢复。
融合 Clip1 使受体的熔解温度(Tm)提高了约 10 °C。
在 80 °C 条件下的配体结合活性保持时间明显延长,表明融合蛋白显著提高了构建的构象稳定性。
冷冻电镜结构解析(Fig.3)
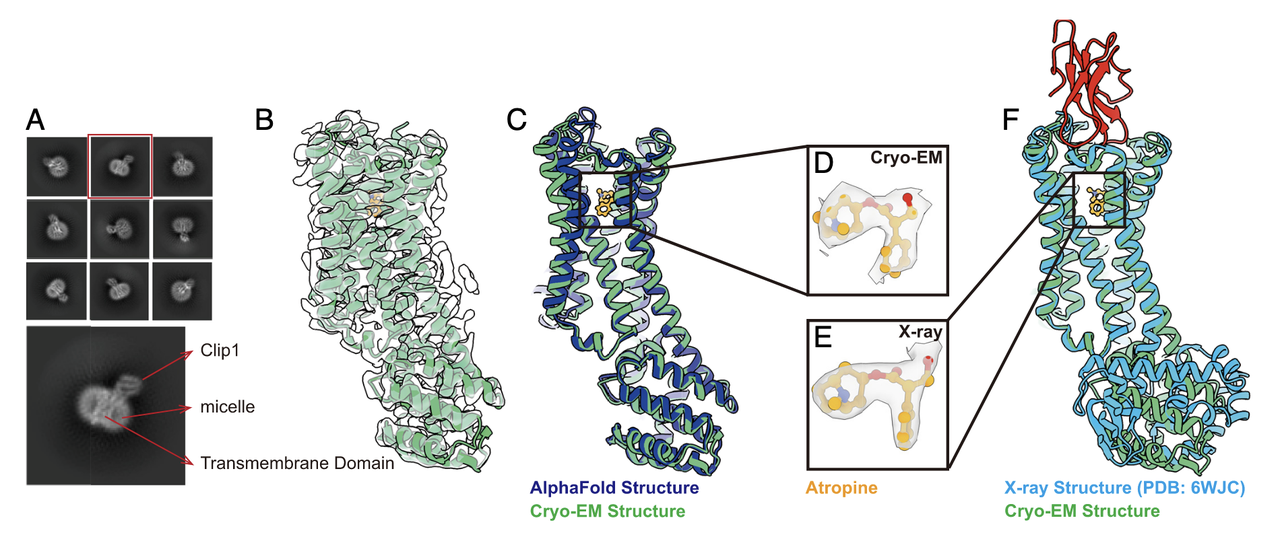
利用 M1R-Clip1 纯化样品进行 cryo-EM 数据采集,仅用约 2,000 张显微图像便获得了分辨率 3.3 Å 的结构重建。
在 2D 分类图像中可清晰识别 Clip1 融合域的特征信号,支持其在粒子对齐中的作用。
cryo-EM 结构与 AlphaFold2 预测模型高度一致,并与此前 X-ray 晶体结构在整体构象及配体结合位点上均无显著差异。
在跨膜螺旋顶部观察到一团未解释的密度,形态类似 atropine,但尚不能确定其来源,有待进一步研究。
小结:M1R 案例验证了 Click Fusion 策略在不破坏受体天然功能的前提下,能够显著提高构象稳定性,并为高分辨率 inactive 状态结构解析提供可靠支撑
4. NMBR 结构解析
在验证了 M1R 的可行性后,作者将 Click Fusion 策略应用于神经降压素 B 受体(Neuromedin B receptor, NMBR)。NMBR 属于 class A GPCR,可介导中枢神经系统及其他组织中的多种生理过程,但长期以来缺乏 inactive 状态的高分辨率结构,这在很大程度上限制了作用机制和药物结合模式的研究。
- 结构获取与整体特征(Fig.4A、B)
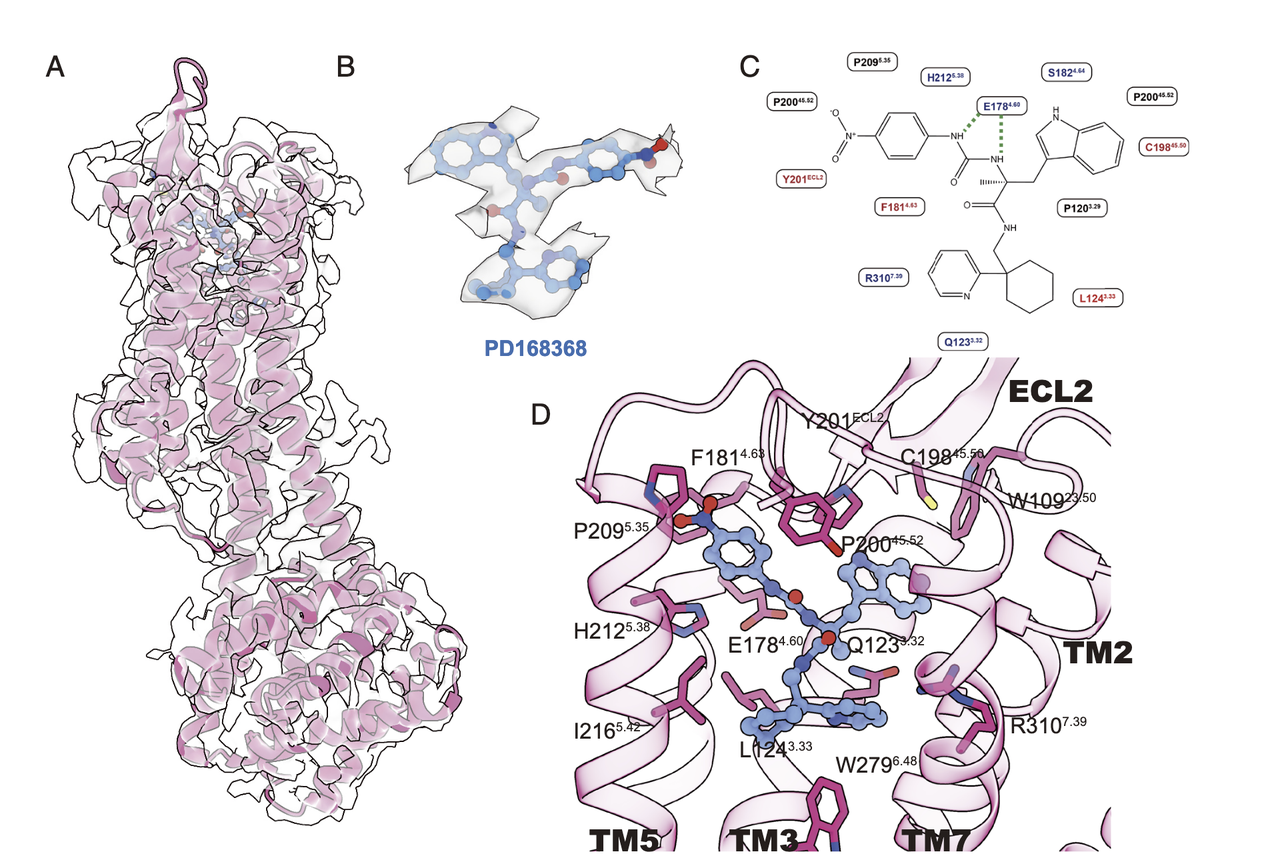
基于 NMBR 的 TM5/TM6 区域设计 Clip2 融合构建(NMBR-Clip2),成功解析了 NMBR 与拮抗剂 PD168368 复合物的 3.4 Å cryo-EM 结构。
cryo-EM 密度图中,拮抗剂在受体正构结合口袋(orthosteric pocket)中呈现出清晰的三叶草形密度,结合构象与预测模型高度一致。
激活与抑制构象差异(Fig.4E)
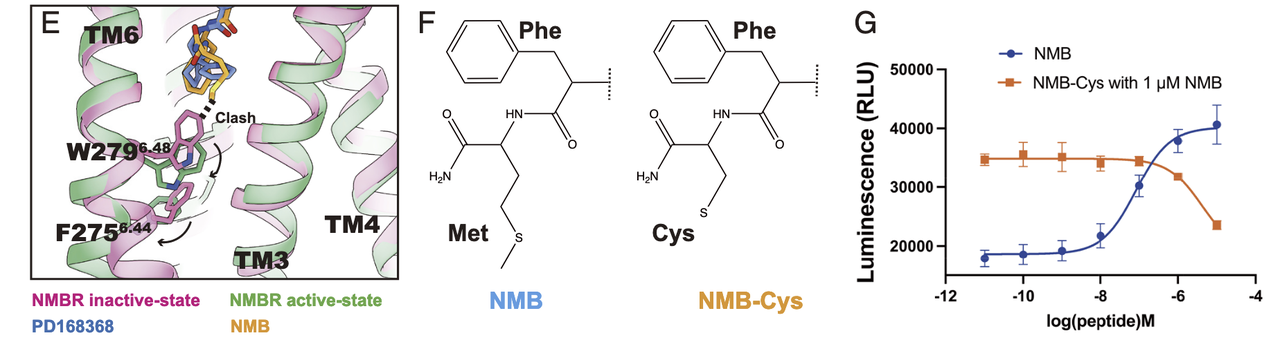
与活化态结构(PDB: 8H0P)比较,inactive 态的 NMBR-Clip2 呈现典型的 TM6 向外位移减小、TM5 向 TM6 偏移的特征。
ECL2(第二细胞外环)在激活过程中向受体核心明显内移,TM1 也发生轻微旋转。
PD168368 占据了正构结合口袋的下部,阻断了激活所需的关键构象变化。
基于结构的功能验证(Fig.4F、G)
作者基于结构推测,NMBR 内源性激动剂 NMB 在 C 端的甲硫氨酸(Met)深入结合口袋,与 W6.48 残基发生空间冲突,推动该“开关”残基向下移动,从而触发受体激活。
将该甲硫氨酸替换为体积更小的半胱氨酸(NMB-Cys)可导致结合更浅,无法推动 W6.48 的构象变化,预期表现为无激活能力。
NanoBiT G 蛋白偶联实验验证了推测:NMB-Cys 无法激活 NMBR,并表现出拮抗作用。
小结:在缺乏已有 inactive 状态结构的 GPCR 中,Click Fusion 策略能够直接获得高分辨率结构,并结合结构信息解释配体的激活或抑制机制,为基于结构的药物设计提供可靠依据。
5. 跨受体迁移测试:5-HT₂BR
为评估 Click Fusion 策略的可迁移性,作者选择了5-羟色胺 2B 受体(5-hydroxytryptamine 2B receptor, 5-HT₂BR)作为验证对象。该受体与纤维化、心血管疾病、肿瘤以及神经系统和胃肠道功能的调节密切相关,也是多种药物开发的重要靶点。
- Clip 的跨受体转移设计(Fig.5A)
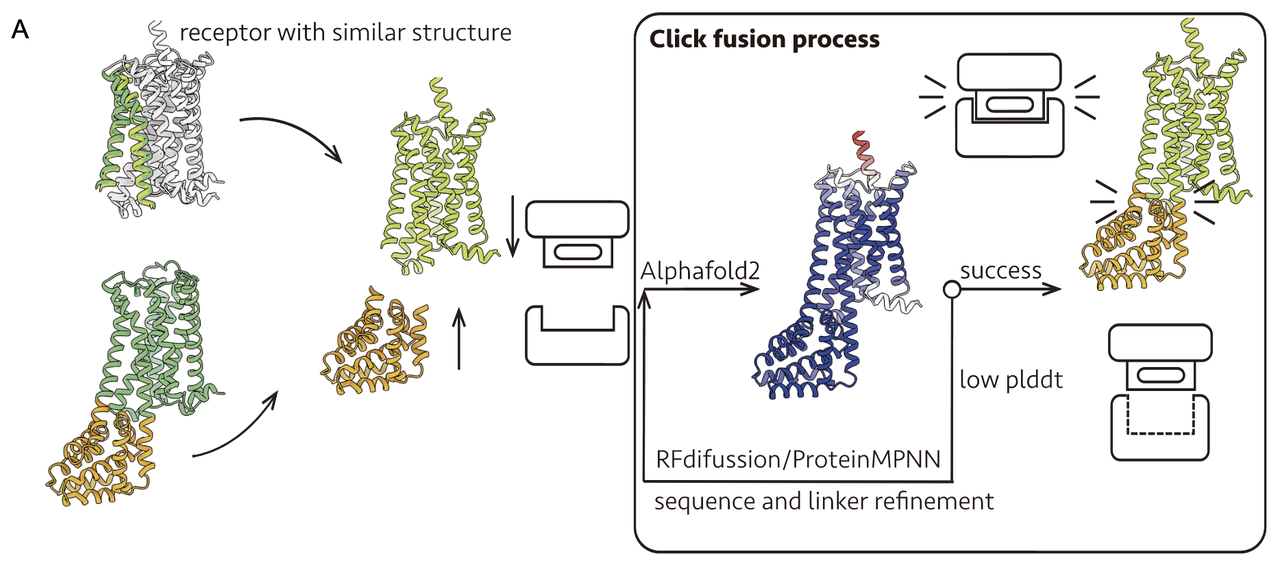
5-HT₂BR 与 M1R 在 TM5/TM6 区域的骨架结构高度相似。作者将已在 M1R 上验证成功的 Clip1 直接迁移至 5-HT₂BR,仅对连接区域进行适当截短和调整。
AlphaFold2 预测模型显示该融合构建在全局及连接区域均具有较高的 pLDDT 评分,表明结构可行性较高。
这一过程不需重新设计骨架,体现了 Click Fusion “即插即用”的优势。
结构解析与配体结合特征(Fig.5B–D)
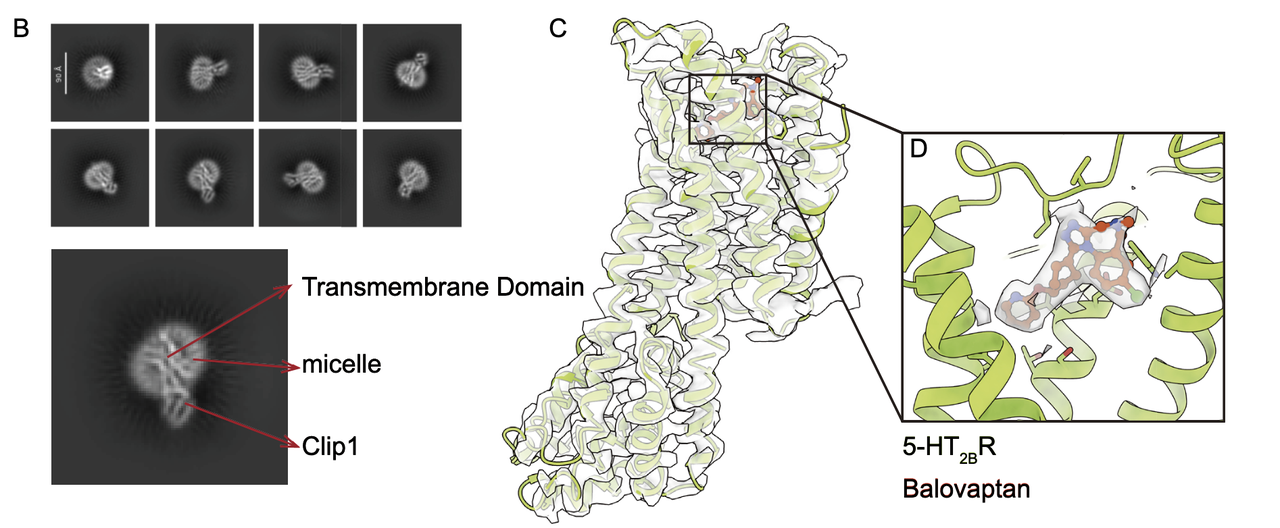
利用 5-HT₂BR-Clip1 与拮抗剂 Balovaptan 复合物进行 cryo-EM 数据采集,最终获得分辨率 3.6 Å 的结构。
正构结合口袋内的 Balovaptan 由三部分结构单元组成:
含氮吡啶环定位于 TM5/TM6 交界面,与多处芳香族残基(F340^6.51、F341^6.52、F217^5.36、A225^5.46)形成疏水或 π–π 堆叠作用;
三环结构单元位于 TM2/TM3 区域附近,嵌入由 L209^4.52、C207^4.50、A111^2.61、W367^7.40、W370^7.43 构成的疏水亚口袋中;
中央环己烷桥连接两端结构,稳定配体构象。
相比其他拮抗剂,Balovaptan 在结合口袋内占据更大空间,可能赋予其独特的结合选择性。
Clip 迁移的适用性与限制
Clip1 在 5-HT₂BR 上表现出良好的表达水平与结构稳定性,迁移成功。
然而,当尝试将 NMBR 上的 Clip2 迁移至 5-HT₂BR 时,融合构建的稳定性和表达均不理想,说明不同 GPCR 的 TM5/TM6 区域结构差异会影响 Clip 的兼容性,需针对性优化。
小结:Click Fusion 具备跨受体迁移的潜力,可在结构相似的 GPCR 间直接复用融合蛋白,从而显著降低构建和优化成本。不过,该方法的适用性仍依赖于目标受体与原始设计受体在融合位点结构上的相似程度。
6. 方法意义与应用前景
本研究通过 de novo 设计的融合蛋白策略,实现了对三种 class A GPCR(M1R、NMBR、5-HT₂BR)inactive 状态结构的解析,并验证了方法的可迁移性和适用范围。与现有稳定策略相比,Click Fusion 具有以下显著优势:
- 高效的 in silico 蛋白工程
大部分构建优化工作在计算机上完成(结构预测、骨架生成、序列设计、刚性评估等),减少了对大规模实验筛选的依赖。
可在短时间内完成从设计到验证的迭代,提高构建效率。
提升受体稳定性
融合 Clip 显著提高了 GPCR 在高温和去配体状态下的构象稳定性(M1R 案例中 Tm 提升约 10 °C),为受体在体外长期保存、纯化及后续实验提供了条件。
这种高稳定性在基于纯化蛋白的药物筛选(如 DNA 编码化合物库、酵母展示、噬菌体展示、核糖体展示等)中尤为重要,特别适用于寻找特异性识别 inactive 状态的配体。
- 结构解析难题的解决方案
针对 inactive 状态 GPCR 缺乏稳定支架的问题,Click Fusion 提供了显著的可识别融合域信号,改善了 cryo-EM 粒子对齐质量。
对缺乏已知 inactive 结构的 GPCR(如 NMBR),该方法能够直接获得高分辨率结构,为药物结合模式解析和作用机制研究提供基础。
- 可迁移性与“即插即用”潜力
在结构相似的 GPCR 间(如 M1R 与 5-HT₂BR)可直接迁移 Clip,无需重新设计骨架,实现快速构建。
虽然迁移性受限于融合位点的结构兼容性,但该策略在 GPCR 家族内部的通用性仍具有较大应用空间。
- 拓展至其他膜蛋白的可能性
- 除 GPCR 外,Click Fusion 的设计理念可推广至小型膜蛋白(如转运蛋白、离子通道等),既可作为结构解析工具,也可作为稳定工程的通用平台。
小结:Click Fusion 将 GPCR inactive 状态的结构解析从高度依赖实验筛选的工程问题,转化为可通过计算快速实现的设计流程,不仅有助于加速拮抗剂药物的结构基础研发,还为膜蛋白稳定性工程提供了新的思路与方法。
7. 点评:
通过将人工智能驱动的蛋白设计与 GPCR 结构生物学相结合,师兄这项研究提出的 Click Fusion 策略为解析 GPCR 的 inactive 状态结构提供了高效、通用的新方案。在 M1R、NMBR 与 5-HT₂BR 三个不同受体的应用案例中,该方法均展现出稳定性提升、结构解析质量改善及一定程度的跨受体迁移能力。
与传统依赖纳米抗体或融合蛋白的稳定策略相比,Click Fusion 最大的创新在于将复杂的构建优化过程前置到计算机端,在设计阶段即可获得高置信度、可直接用于实验的构建方案。这不仅显著减少了实验筛选的成本与周期,也为 GPCR 结构解析和基于结构的药物设计开辟了新的技术路径。
在 GPCR 占据药物靶点重要地位的背景下,Click Fusion 有望在拮抗剂药物研发、受体稳定性工程以及膜蛋白结构解析等多个领域发挥作用。未来,随着蛋白设计工具的不断优化,该策略的适用范围有望进一步扩展至其他类型的膜蛋白,为结构生物学和药物化学提供更丰富、更高效的技术支持。
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →