从结构出发设计肽药,BindCraft 可行吗?

关键词:Peptides, Alphafold, BindCraft, De novo peptide design
🔗原文链接:https://www.biorxiv.org/content/10.1101/2025.07.23.666285v1
研究总结
这篇论文《Generative Design of High-Affinity Peptides Using BindCraft》主要研究了使用一个叫 BindCraft 的计算工具来“从蛋白质结构出发,生成高亲和力的肽类配体(peptide binders)”。
研究目标
作者希望评估 BindCraft 是否能:
从目标蛋白的三维结构出发,自动生成能特异性结合的短肽序列(10–20个氨基酸);
为药物开发提供一个无需复杂筛选实验的替代手段。
研究方法与主要实验
- 工具简介
BindCraft 是一个基于 AlphaFold 的结构引导生成模型,原本设计用于生成“迷你蛋白(miniprotein)”结合物。这篇论文尝试将其用于生成更短的、潜在更具药用价值的“肽类”。
两个目标蛋白:MDM2 和 WDR5
MDM2:一种癌症相关蛋白,天然与 p53 蛋白结合。
WDR5:与染色质修饰和癌基因如 MYC 有关,具有两个已知的结合位点:WIN 和 WBM。
生成与实验验证
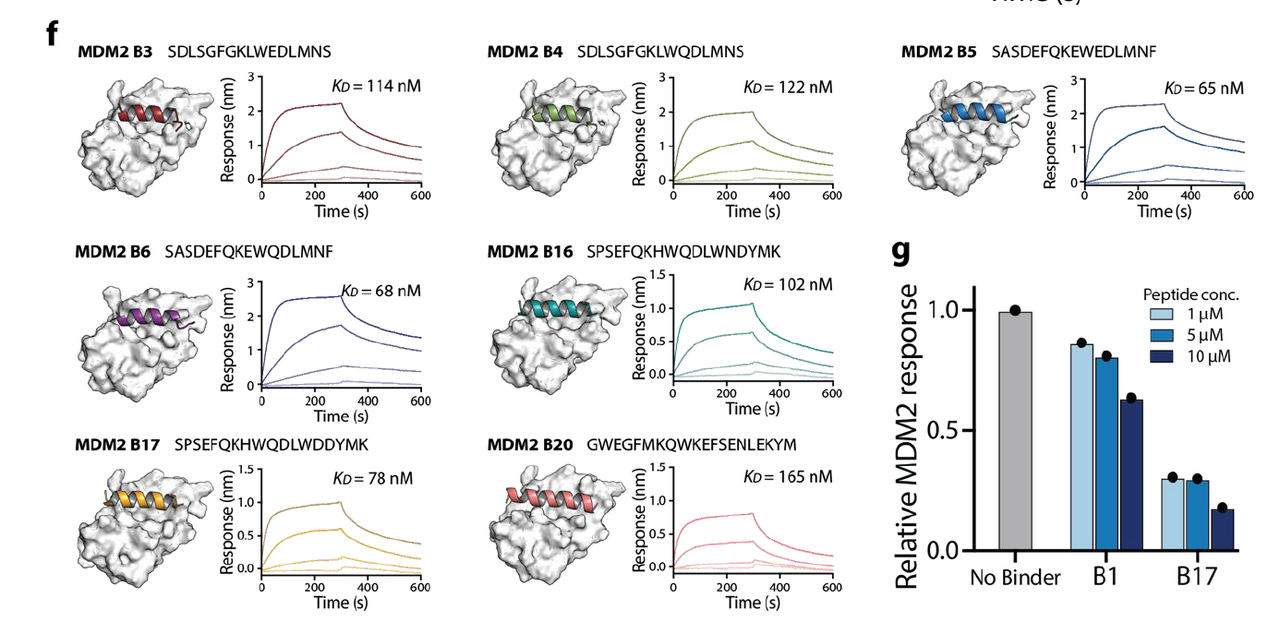
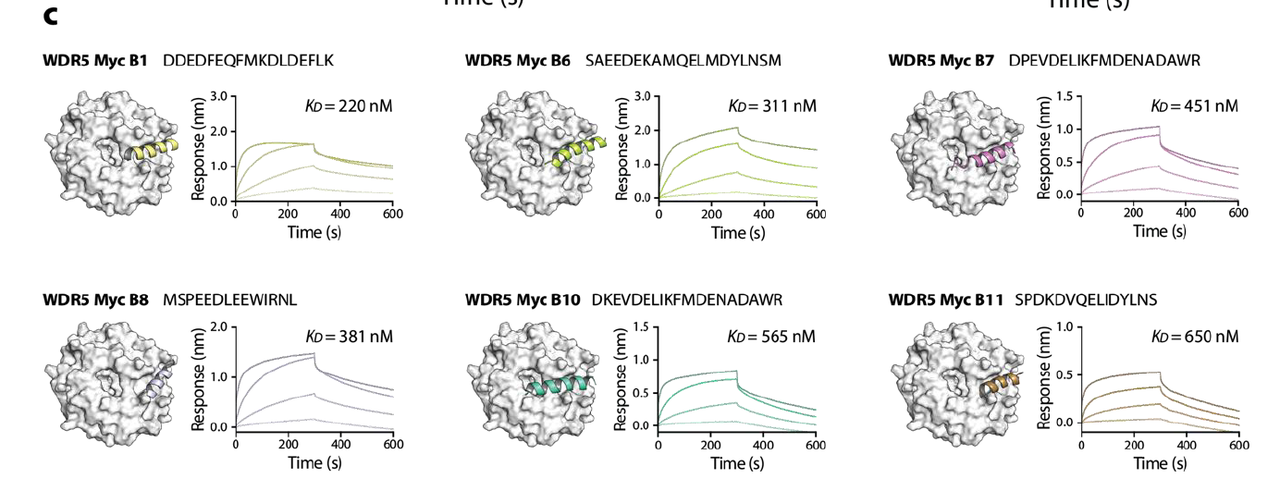
BindCraft 生成了针对 MDM2 的 70 条候选肽链,其中合成了 15 条,有 7 条显示出纳摩尔级别的结合亲和力(KD = 65–165 nM)。
对 WDR5 的两个结合位点分别生成了肽,其中 WIN 位点无有效结合肽,但在 WBM 位点发现了 6 条 KD 在 219–650 nM 的结合肽。
进一步对最佳肽做了“Stapling(主链钉固化)”结构修饰,使其稳定 α-螺旋结构,结合力提升到 KD = 39 nM。
研究结论
BindCraft 可以用于从蛋白结构中自动生成功能性肽类结合剂,具备一定的命中率。
成功生成 MDM2 和 WDR5(WBM位点)的高亲和力肽,失败于 PD-1/PD-L1 和 WDR5 WIN 位点。
相较于 phage display 等实验筛选方法,BindCraft 提供了结构信息,可直接用于优化,如肽钉固。
对于不具备深度计算背景的实验室,BindCraft 是一个“可直接上手”的工具。
一、研究背景:为什么要“从结构出发设计肽药”?
现代药物开发越来越重视蛋白–蛋白相互作用(PPI)的干扰,而肽类分子是阻断这类接口的理想形式。相比小分子,肽能更好地覆盖大而平坦的蛋白结合面;相比迷你蛋白(miniprotein),肽具有更好的合成性、药代属性和潜在的细胞穿透性。
然而,如何快速找到能结合目标蛋白的高亲和力肽,一直是药物设计中的难题。传统的筛选手段如 phage display 或 mRNA display 成本高、周期长,且不能主动控制靶点结构或结合位点。
于是一个关键问题被提出:
“我们能否直接从蛋白质三维结构出发,通过计算工具自动生成具有结合能力的短肽序列?”
这正是 BindCraft 平台所尝试解决的问题。
二、技术工具:什么是 BindCraft?
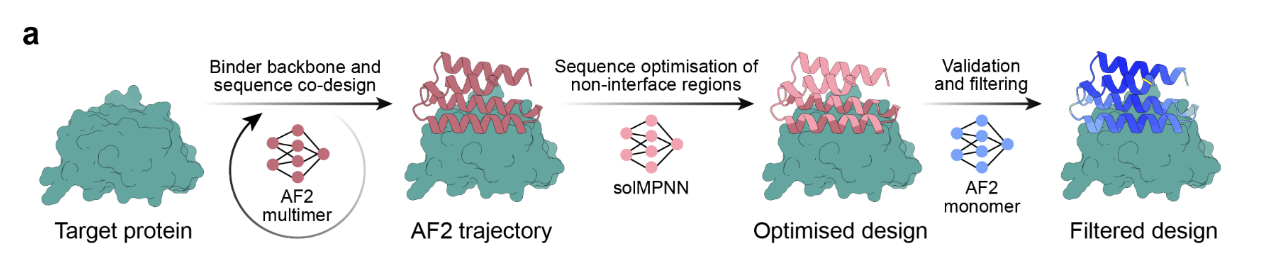
BindCraft 是一个基于 AlphaFold 架构开发的结构引导生成模型,原本用于生成小型蛋白(miniproteins)结合配体。它的主要特点包括:
输入:目标蛋白结构(PDB)+ 目标结合位点残基;
输出:一批结构上拟合结合界面的肽或小蛋白序列;
附加输出:预测的肽-蛋白复合物结构,可用于后续优化;
目标用户:没有计算背景的药物/化学/结构实验室也可使用。
研究者希望通过这项研究来评估:
“BindCraft 能否也用于短肽(10–20 个氨基酸)的结构引导生成?”
三、第一步:以 MDM2 为模板的“概念验证”
为什么选择 MDM2?
MDM2 是一个非常经典的肽结合靶点:
它与 p53 蛋白结合界面小且明确;
结构清晰(PDB: 1YCR);
天然结合肽是短 α-螺旋结构,含关键疏水三元组 F/W/L;
已有大量肽类优化数据作对比。
BindCraft 如何操作?
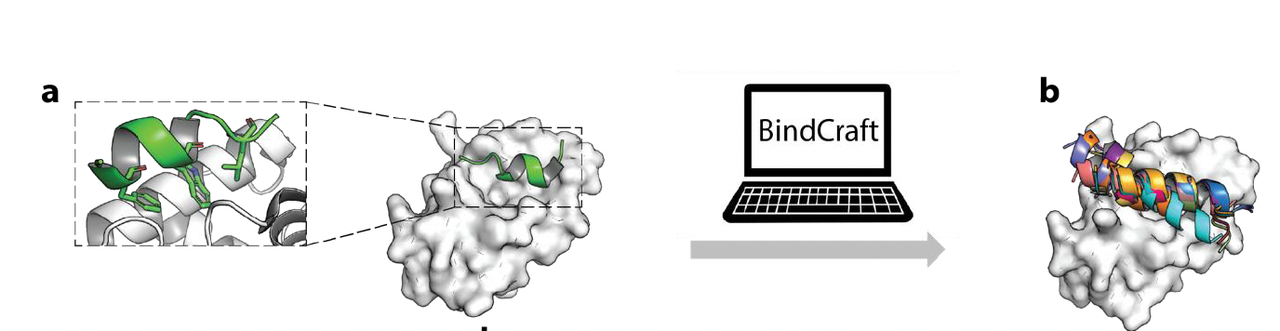
研究者将 MDM2 的结构输入 BindCraft,并指定 p53 结合界面关键残基作为目标区域,生成了 70 条候选肽(10–20 个氨基酸)。
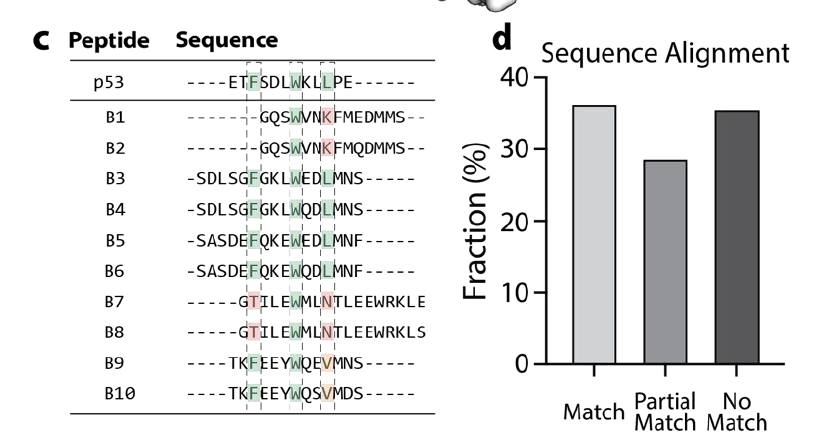
结构分析结果:
所有候选肽预测结构均为 α-螺旋;
其中约 36% 含有完整的 F/W/L 热点三元组;
另有 28% 包含部分热点(如 F/W/V、F/M/L 等)。
实验验证:
合成了其中的 15 条;
使用 BLI 测定与 MDM2 的亲和力,有 7 条表现出清晰的结合动力学(KD = 65–165 nM);
大多数有效肽中确实包含 F/W/L;
通过竞争实验验证这些肽能阻断 p53 的结合,说明靶点位点是正确的。
✅ 阶段性结论:
BindCraft 可以从结构出发生成高命中率的短肽候选,其中包含已知关键结合元素,并且实验上可验证其功能活性。
四、第二步:从“简单靶点”转向“结构复杂”的 WDR5
为什么选择 WDR5?
WDR5 是另一个重要的致癌蛋白,具有两个已知的结合界面:
WIN 位点(与 MLL 蛋白结合);
WBM 位点(与 MYC 蛋白结合)。
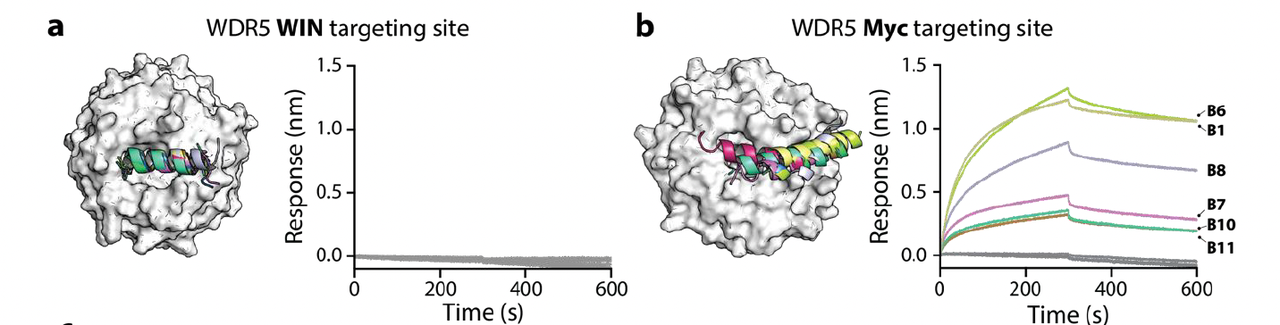
与 MDM2 不同的是:
两个位点都不是天然 α-螺旋结合;
天然肽为 loop 或柔性结构;
是对 BindCraft 的一次挑战。
实验设计:
对两个位点分别进行 BindCraft 设计:
WIN site:以 PDB 6DY7 中的 F133 为锚点;
WBM site:以 L240 为锚点;
各生成 100 条肽,选前 10 条合成验证。
实验结果:
WIN site 所有肽均无结合活性;
WBM site 中有 6 条肽显示出中等亲和力(KD = 219–650 nM);
有效肽中普遍富含带负电残基,与 WDR5 的正电结合面匹配。
❌ WIN失败,WBM成功。
✅ 阶段性结论:
BindCraft 可能偏向于设计 α-螺旋结构的结合肽,这对于 WIN site 等“柔性结合界面”是挑战;但在 WBM site 上仍能生成功能性肽,展示其适应不同结构位点的潜力。
五、第三步:结构驱动的优化 —— Stapling 案例
在 WBM 成功肽中,研究者选取 Myc_B1 做结构优化,验证 BindCraft 输出结构的实际应用价值。
设计思路:
- 利用预测结构识别两个不参与结合、背面暴露的残基 Glu6 和 Lys10;
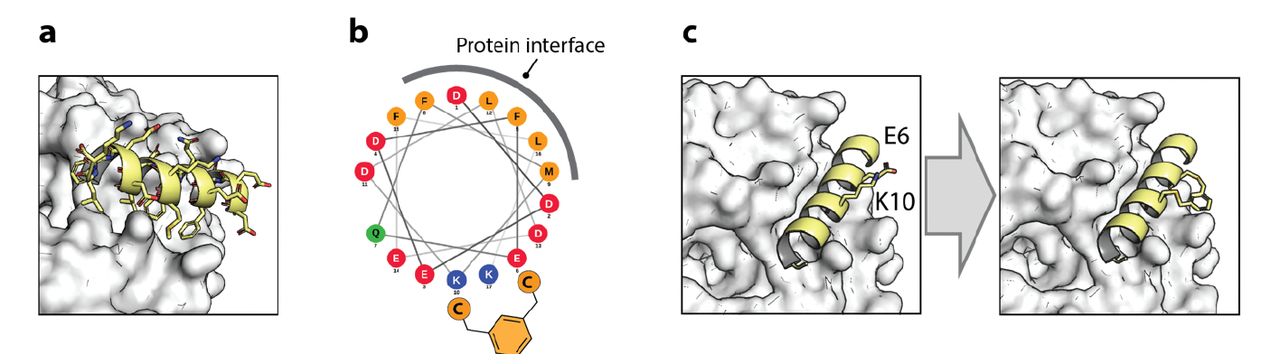
- 将其换成半胱氨酸,用 meta-xylene 做 i,i+4 的 Stapling;
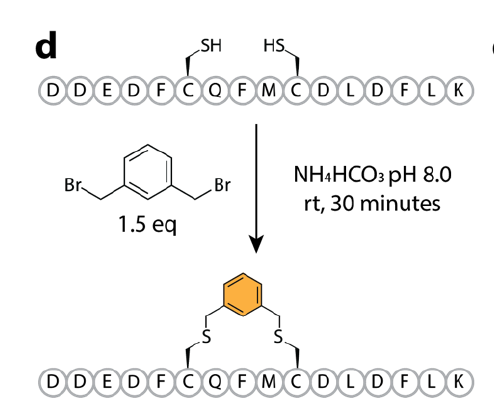
- 测定其亲和力与功能抑制效果。
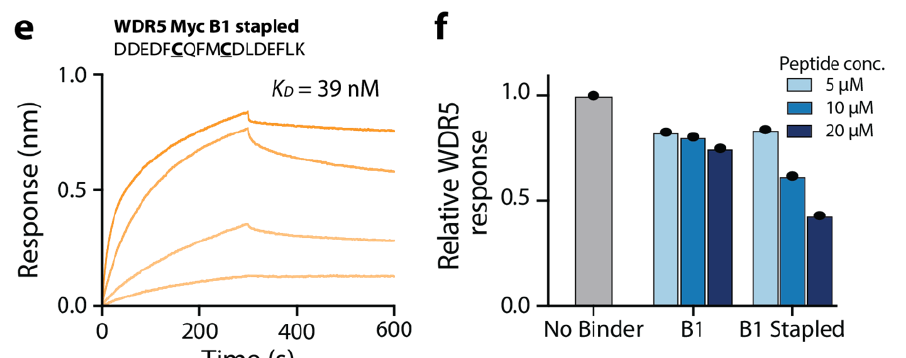
实验结果:
Stapled 版 KD = 39 nM,比原始版本高 6 倍;
在竞争实验中,stapled 肽能更强地抑制 WDR5 与 MYC 的结合(最大抑制 57% vs 25%)。
✅ 阶段性结论:
结构预测不仅指导 hit 设计,还能直接用于药化优化。传统 display 技术不具备这种“结构优化”的路径,显示了 BindCraft 更强的后期扩展性。
六、总体评价与研究贡献
✅ 主要发现:
BindCraft 可生成功能性短肽 binders,尤其适用于 α-螺旋结合靶点;
成功应用于两个癌症相关蛋白(MDM2 和 WDR5);
生成肽具备高度结构合理性,可直接进行优化设计(如 stapling);
使用门槛低,无需修改代码,适合普通实验室操作。
⚠️ 局限性:
对于非 α-螺旋结合界面(如 PD-1/PD-L1, WDR5-WIN),设计命中率较低;
可能内在偏好螺旋类结构,限制某些接口设计能力。
🧩 结语:从肽的结构生成到功能验证,BindCraft 展示了“结构驱动型药物设计”的新范式
这项研究不是简单地展示了一个 AI 工具的性能,而是围绕“结构–设计–验证–优化”构建了一整条药物早期开发路径的雏形。对于希望探索“计算辅助肽药设计”的研究者来说,它提供了技术路径、成功模板和挑战边界,是一篇具有代表性的里程碑式研究。
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →