AlphaGenome:基因组学的 AlphaFold 时刻?

现代基因组学面临的最大挑战之一,是如何解释 非编码区域的遗传变异。这些变异占据了我们基因组的98%以上,但却不直接编码蛋白质——它们调控的是“什么时候、在哪里、如何”表达基因。
过去,研究人员通常需要多个专用模型来分别预测:
剪接(SpliceAI、Pangolin)
染色质可接近性(ChromBPNet)
表达调控(Borzoi)
3D结构(Orca)
但这种碎片式的方式不仅繁琐,还限制了全局调控机制的解析。
基于此,昨天(2025年6月25日)DeepMind 提出了 AlphaGenome:一个统一建模的AI系统,能一次性预测11种基因调控模态,兼顾远程调控建模能力和单碱基分辨率。这就是本文的主角。

一、AlphaGenome是什么?一个把整个基因调控“装进一个模型”的系统
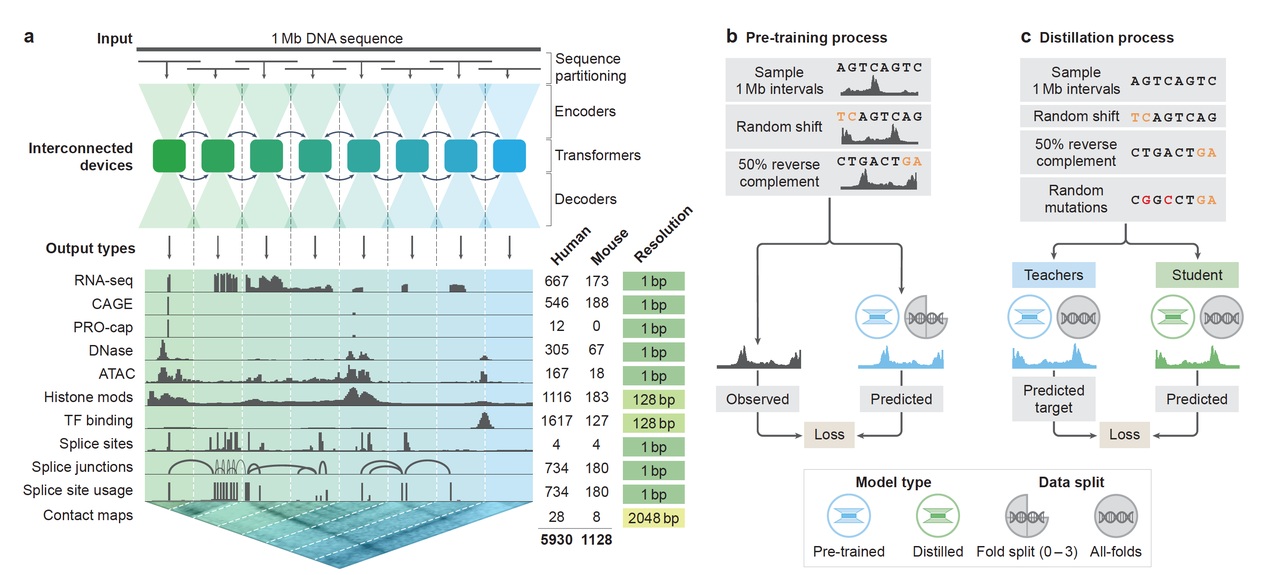
AlphaGenome 输入的是长达 1 Mb(百万碱基) 的 DNA 序列,通过 U-Net + Transformer 模型架构进行处理,输出 11 类功能轨迹,涵盖:
| 模态类型 | 示例数据 |
|---|---|
| 转录表达 | RNA-seq、CAGE、PRO-cap |
| 剪接机制 | 剪接位点、剪接连接、使用率 |
| 染色质可及性 | DNase-seq、ATAC-seq |
| 表观遗传修饰 | 各类ChIP-seq(如H3K27ac) |
| 转录因子结合 | TF ChIP-seq |
| 三维染色质结构 | Hi-C/micro-C contact maps |
它采用两阶段训练策略(图1b–c):
预训练(多个教师模型),学习全基因组各模态的实验数据。
蒸馏成学生模型:只需一次推理,即可预测所有模态和变异效应,不到1秒/个变异。
这让AlphaGenome具备多模态、高分辨率、长范围建模、推理高效等综合能力。
二、AlphaGenome准吗?全面性能碾压所有主流模型
在 24 个基因轨迹预测任务中,AlphaGenome 在 22 个上表现最优,大多数模态相对提升 10%–40% 不等(图1d)。
在 26 个变异效应预测任务中,24 个任务达成SOTA(图1e),包括:
剪接变异(超越 SpliceAI、Pangolin)
表达变异(eQTL、GTEx:超越 Borzoi)
染色质状态变异(超过 ChromBPNet)
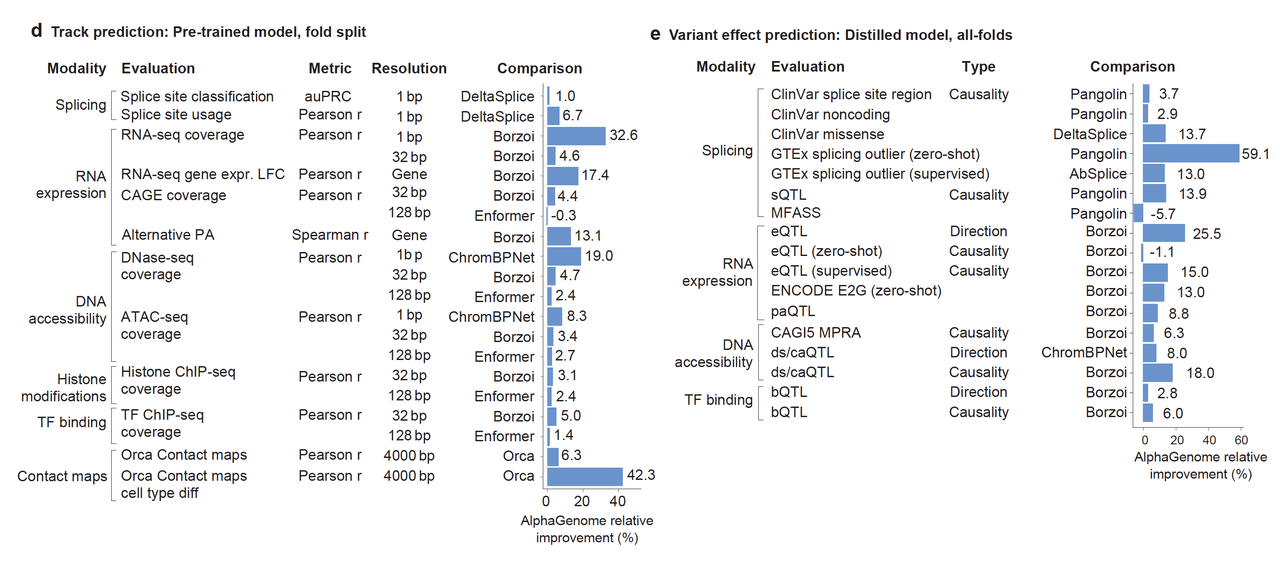
AlphaGenome不是某个任务做得好,而是全方位做得好。
三、AlphaGenome怎么做到的?深入案例剖析剪接、表达、染色质预测
A. 剪接预测:不是“位点预测器”,而是“剪接建模机”
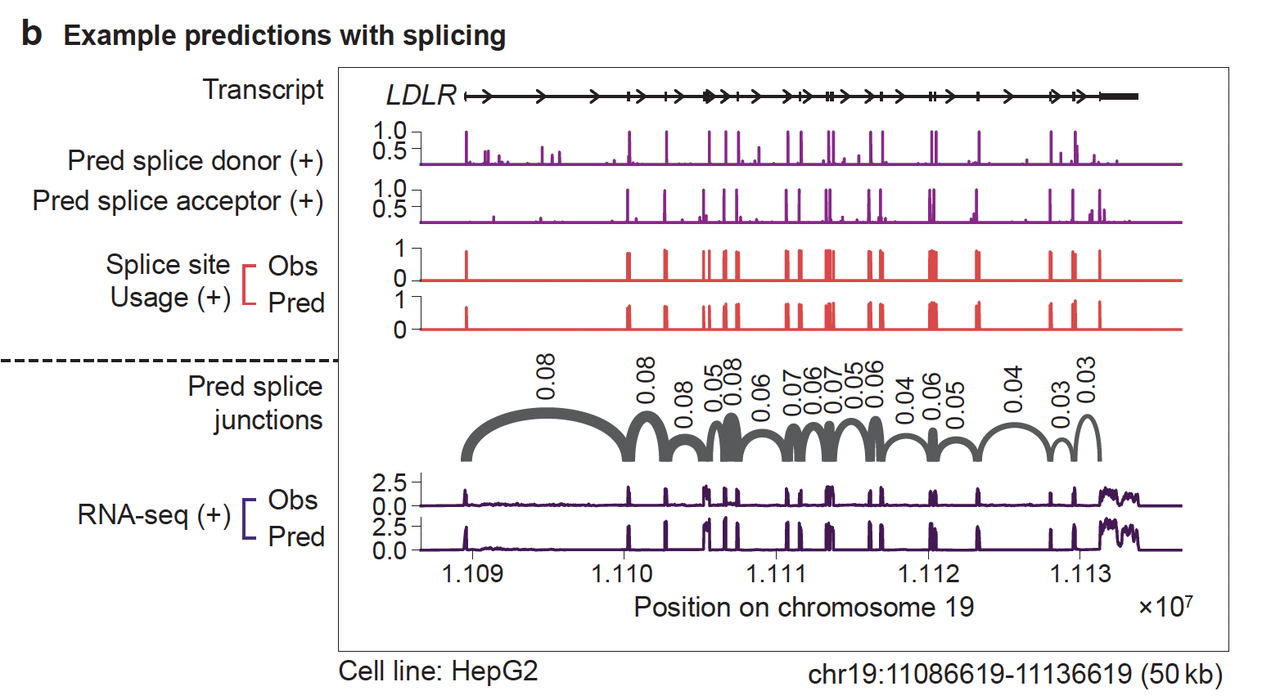
AlphaGenome 是唯一同时预测:
剪接位点(donor/acceptor)
使用率(usage)
剪接连接(splice junctions)
RNA-seq 跨外显子的 coverage 变化
的计算工具,其他的工具都只能预测其中一个或两个任务。
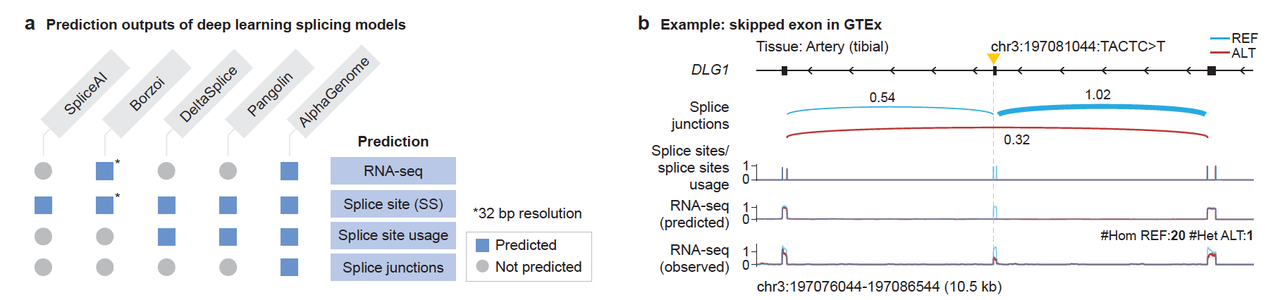
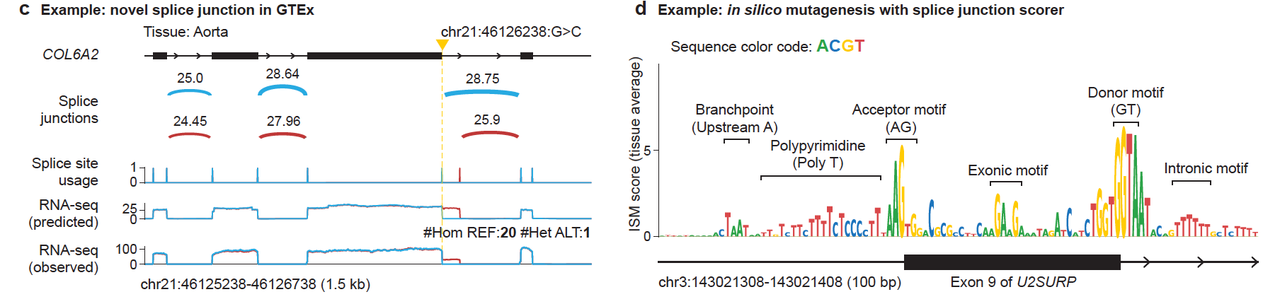
在多个 GTEx 剪接异常的案例中(图3b–c),AlphaGenome 完全还原外显子跳跃或新剪接连接的形成,且能识别motif机制(图3d)。
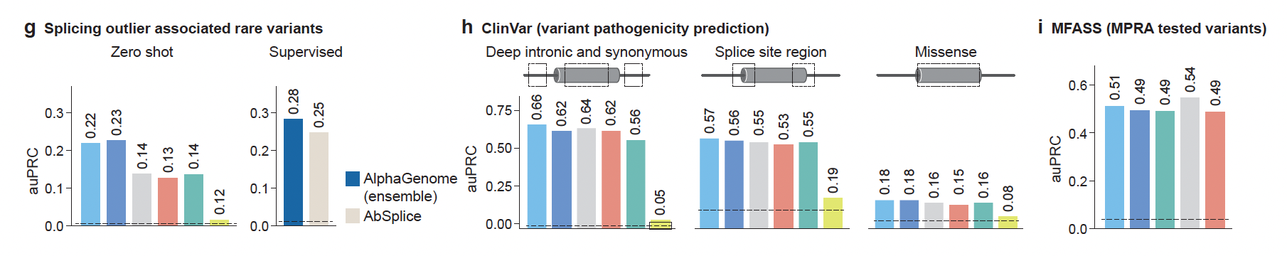
它还在 6 个剪接相关基准任务中拿下冠军,包括 ClinVar 病理剪接变异、GTEx 稀有变异、sQTL、MFASS 高通量验证(图3f–i)。
从预测“哪儿剪”到预测“怎么剪”
🔬 B. 表达预测:不仅预测“强度”,还预测“方向”和“机制”

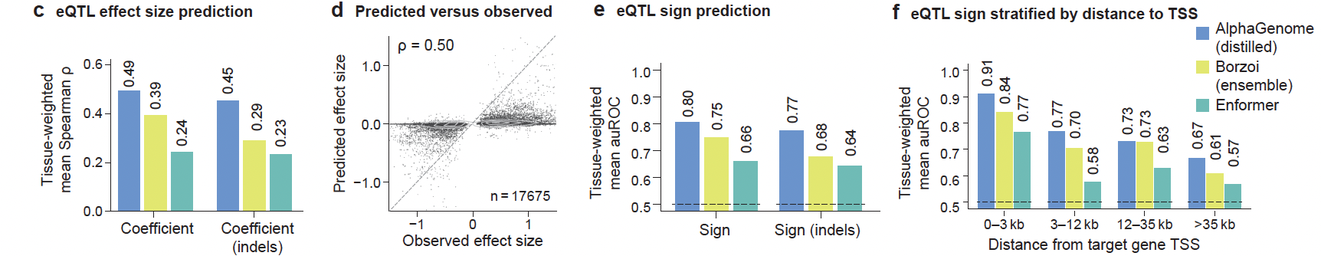
通过在 GTEx 的数据中比较模型预测和实际观测,AlphaGenome 对表达变异(eQTL)预测:
效应强度相关性从 0.39 → 0.49
方向预测准确率 AUROC 提高至 0.80
特别对远距离变异 >35kb 仍有优势(图4c–g)
更关键的是,它能解释 GWAS 信号的方向(图4h)——你知道某个变异与疾病有关,但你不知道它是上调还是下调某个基因?AlphaGenome 能给出答案。
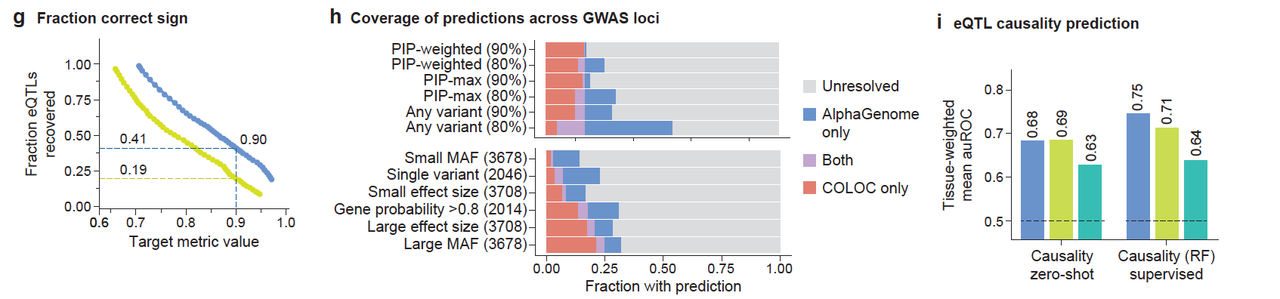
eQTL 信号“定向器”
🔬 C. 染色质与转录因子结合预测:看懂调控开关的“通断”
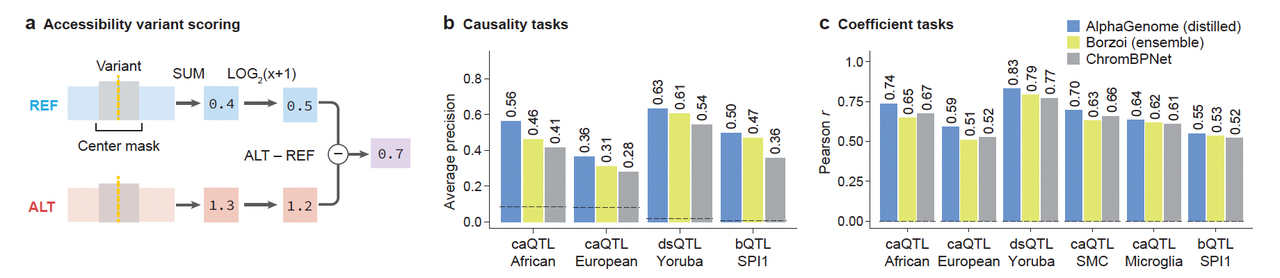
AlphaGenome 在 ATAC、DNase、TF-ChIP 数据上对变异预测也超越了当前强模型:
可接近性 QTL(caQTL)、DNase 敏感性(dsQTL)
转录因子结合 QTL(bQTL)
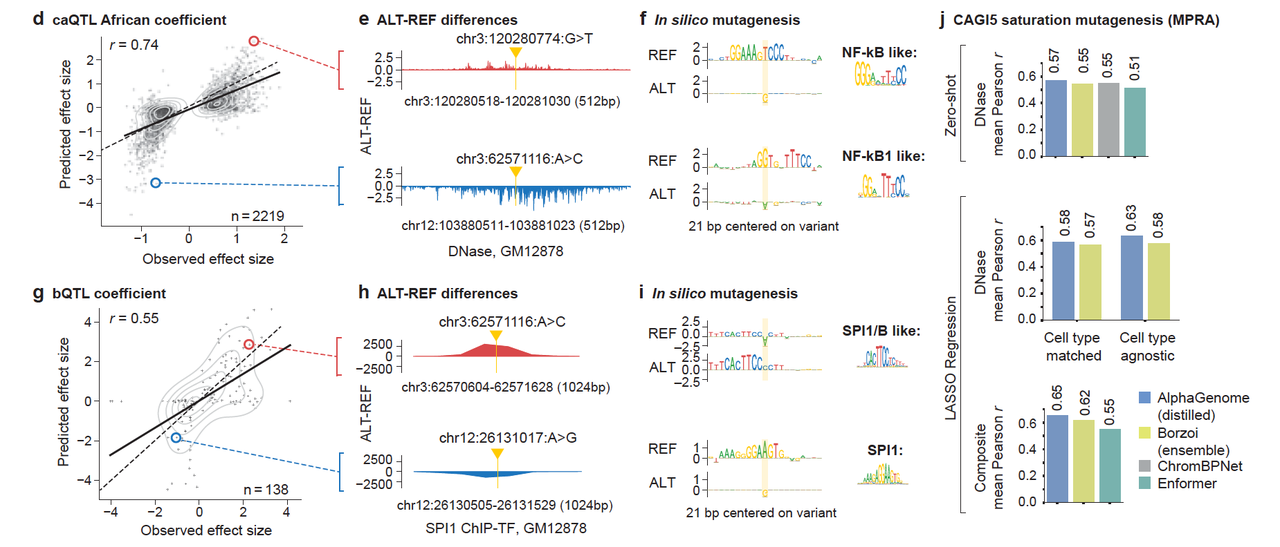
在图5d–i中,模型能精准捕捉 NF-κB、SPI1 等 TF 的结合motif,并通过 in silico mutagenesis 显示哪些核苷酸对绑定最关键。
TF调控“微操控”解释器
四、AlphaGenome能干什么?跨模态解释肿瘤突变机制
在 T-ALL 白血病中,研究者识别了多个非编码致癌突变影响 TAL1 基因的表达。
AlphaGenome 展示了对这些突变的跨模态解释能力:
在突变位点预测出增强子激活(H3K27ac 上升)
TAL1表达预测显著上调
motif分析发现变异引入 MYB motif,为增强子形成机制(图6b–e)
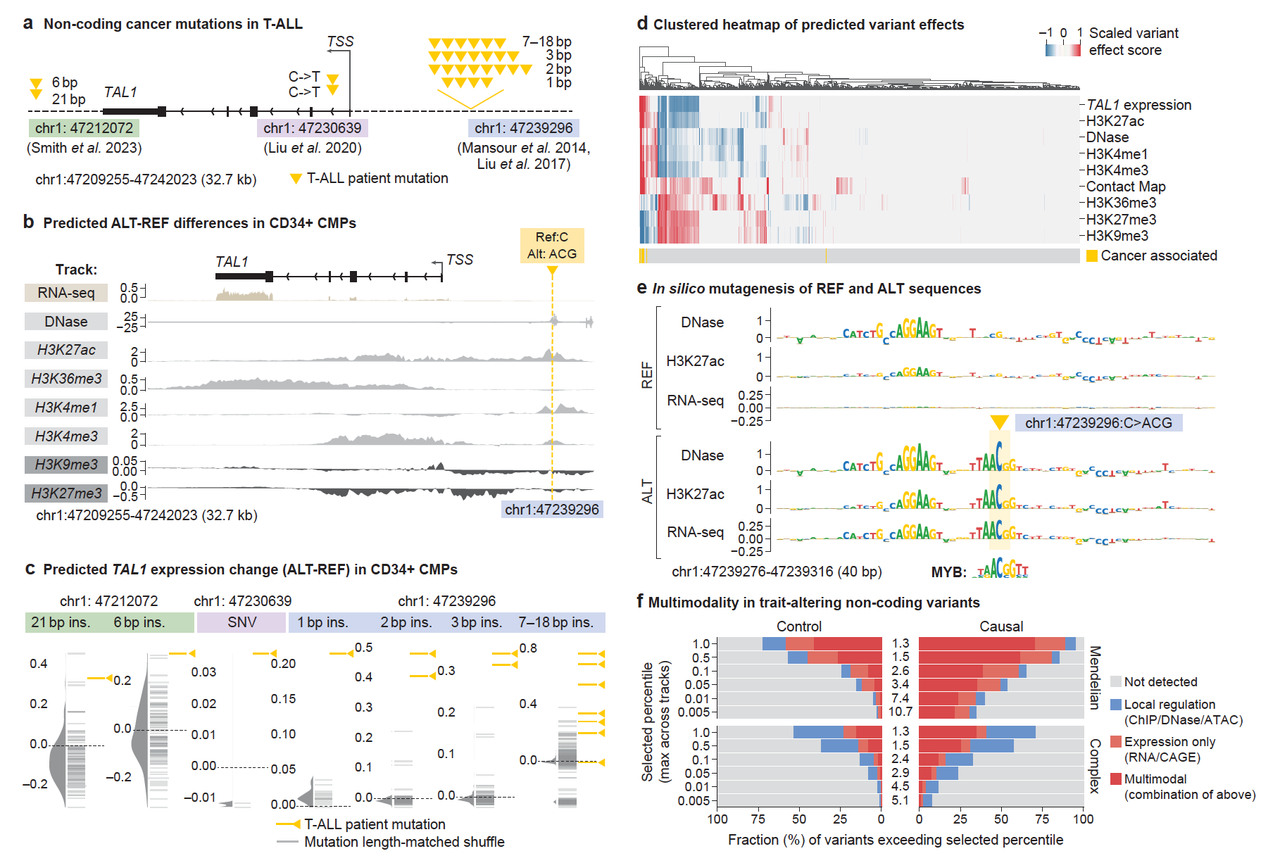
致病突变的“调控解释器”
五、总结:AlphaGenome,不只是“预测器”,更是“调控理解器”
AlphaGenome 不只是像传统的深度学习模型一样,做“分类任务”或“回归任务”那么简单,它是一个能理解调控结构、解释变异机制、支持多任务推理的基因组AI系统。
它的突破来自:
全模态建模
长距离依赖捕捉
高分辨率细节识别
实用的推理效率
做到大一统(全模态)以及具有一定的可解释性,让这个工具的可能发挥的价值无法估量。
🧭 讨论:通用基因组AI的可能与挑战
AlphaGenome 的发布标志着一个关键的转折点:我们正在从零散、模态特异的模型生态,走向一个统一、通用的预测框架。这不仅提升了预测的精度,也重构了我们与基因调控数据之间的交互方式。
一体化预测是必然趋势
传统方法往往依赖“一个任务一个模型”:
剪接用 SpliceAI
开放性用 ChromBPNet
表达用 Enformer/Borzoi
这种方法虽然精确,但代价是复杂、效率低、无法跨模态整合。而 AlphaGenome 的一体化框架提供了一个更高层次的解决方案,能够:
同时预测多个调控模态
支持单碱基分辨率和远程调控上下文
用一次推理,完成变异效应的全景评估
这不仅在科研中实用,也让大型人类变异数据库的解释变得更加可扩展。
模型能“解释”变异,而不只是“预测”它
AlphaGenome 不仅提供预测分数,还能解释变异背后的调控机制。例如在 TAL1 的白血病案例中,它识别出了变异引入的 MYB motif、染色质状态的改变,以及表达水平的变化——这使模型真正从“预测器”演进成了“调控解释器”。
此外,in silico mutagenesis、剪接连接建模、表达方向预测等功能,也都指向同一个目标:理解变异是如何改变生物系统的。
挑战仍在:组织特异性和机制细节
尽管 AlphaGenome 表现出色,但它并非完美无缺。论文中也坦诚指出:
某些组织特异的表达或剪接模式仍难以精准建模
某些中间层机制(如RNA稳定性、翻译效率)未能显式建模
某些模态(如polyadenylation)预测尚依赖间接信号,未直接训练
这些问题提醒我们,当前的 AI 模型虽强,但在面对极端稀有变异、复杂组织上下文和多因子交互时,仍需更多数据、更丰富的结构和更强的生物归纳能力。
AlphaGenome 启示了什么?
它让我们看到了一个可能的未来:
研究者只需输入一段 DNA 序列或一个变异,即可获得关于该序列在不同生物层面(表达、剪接、染色质、TF绑定等)的影响预测,甚至机制解释。
这不仅节省了模型选择、数据整合的负担,更重要的是,它加速了我们对人类非编码基因组的系统理解。
如同 AlphaFold 改变了结构生物学,AlphaGenome 正在悄然重塑我们解读调控基因组的方式。
博客链接:https://deepmind.google/discover/blog/alphagenome-ai-for-better-understanding-the-genome/#:~:text=Today%2C%20we%20introduce%20AlphaGenome%2C%20a%20new%20artificial%20intelligence,a%20wide%20range%20of%20biological%20processes%20regulating%20genes.
预印本文章链接:https://storage.googleapis.com/deepmind-media/papers/alphagenome.pdf
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →