AI生成可复活噬菌体基因组:Evo模型开启生成式基因组学时代
今天要分享的,是一篇我认为保底CNS 正刊的工作:《Generative design of novel bacteriophages with genome language models》。作者是Arc Institute的Brian Hie,是近年来在 AI 与生物交叉 领域迅速崛起的年轻学者。他的团队 Hie Lab 致力于把机器学习、特别是语言模型应用到分子和基因组的设计中,从蛋白质与抗体优化到整个病毒基因组的生成,都有重要成果。他们的研究目标不仅是理解自然进化的规律,更是利用 AI 加速甚至超越进化,在确保安全可控的前提下,为药物开发和抗菌治疗打开全新路径。



作者团队在这篇文章中利用大型基因组语言模型(Evo),首次实现了 完整噬菌体基因组的生成、合成与复活。
他们不仅证明 AI 可以“写出能活的基因组”,还展示了这些 AI 病毒在多个维度上 超越了天然病毒:
进化新颖性:生成的基因组与已知噬菌体差异巨大,但依旧保持合理的功能框架;
实验验证:302 个候选中有 16 个复活为可感染、可扩增的噬菌体;
性能优势:部分 AI 病毒在适应性、裂解效率上超过野生型 ΦX174;
应用前景:在耐药细菌面前,AI 病毒“鸡尾酒”能突破耐药屏障,而天然 ΦX174 完全失效。
这不仅是噬菌体研究的一次突破,更是 生成式基因组学(generative genomics) 的首次落地。
它标志着 AI 已经不止于蛋白质设计,而是真正迈向了 生命系统的整体设计。
🔗原文链接:https://doi.org/10.1101/2025.09.12.675911

一、背景介绍
如果说“蛋白质设计”是近年来合成生物学的热门话题,那么“基因组设计”则是更高阶的终极挑战。
蛋白质不过几百上千个氨基酸,但一个基因组往往包含上千个基因、无数调控元件和复杂的互作关系。一个点突变可能就让整个系统崩溃。
在文章的 Figure 1A 中,作者用一张示意图直观展示了基因组设计的复杂性:基因方向性、调控序列、结构域之间的兼容性……每个因素都像齿轮一样咬合在一起,一旦出错,整个系统就可能“报废”。
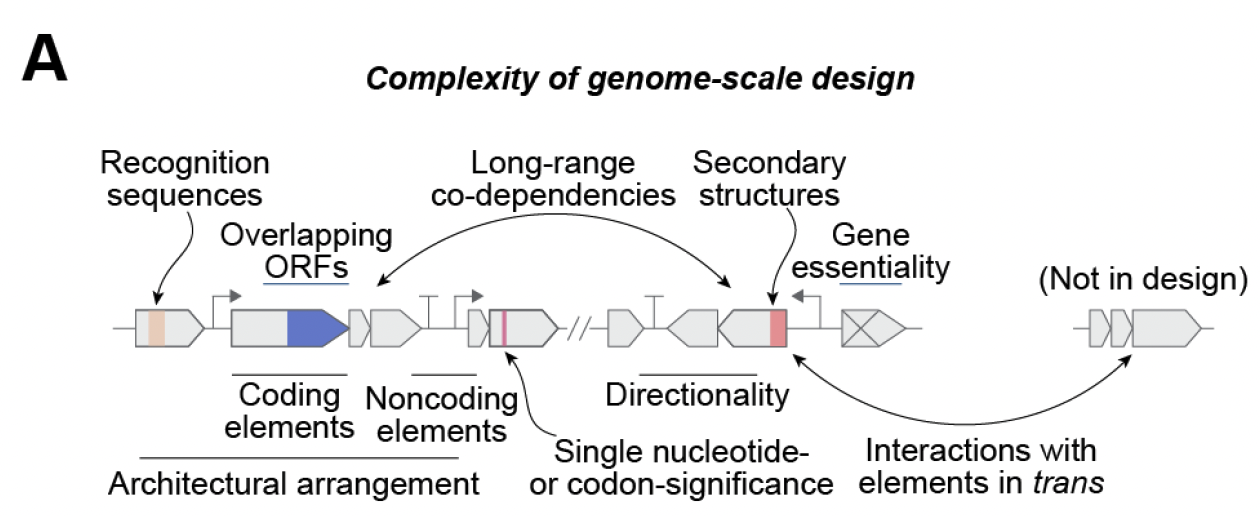
也因此,设计基因组一直被认为是合成生物学的“圣杯问题”。过去几十年里,人类依靠理性设计、随机突变或定向进化,虽然也能改造基因组,但往往局限在局部优化。真正“从零生成”一个全新的基因组,在这篇文章之前一直被视为不可企及。
二、AI 的思路:从自然进化到“语言模型”
自然进化用了几十亿年,不断试错,才塑造出我们今天看到的多样生命。那么,如果我们能把这种“进化的语法”学会,是否就能反向写出新的生命?
作者的思路正是如此:把基因组看作一种语言。
语言模型如何理解基因组?
就像人类语言有语法、语义和上下文依赖,基因组也有自己的规则:哪些碱基可以排列在一起,哪些基因要协同表达,哪些调控元件必须同时存在。
团队训练的 Evo 1 和 Evo 2 基因组语言模型,在超过两百万份噬菌体基因组上进行了大规模预训练,相当于读完了整个“病毒语料库”。
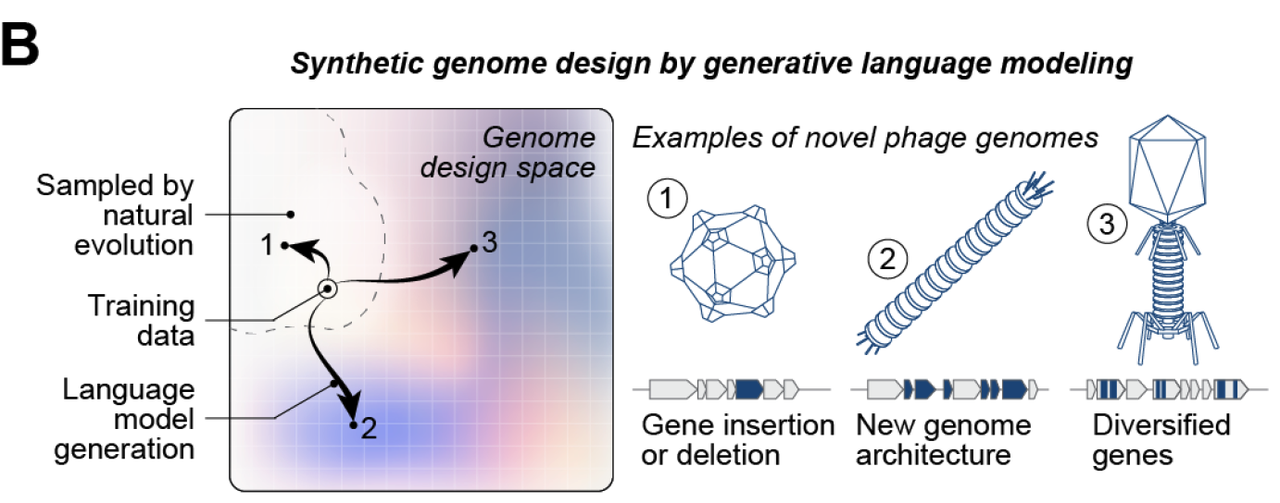
- 在 Figure 1B 中,作者用示意图强调:自然进化只探索了巨大基因组空间的一小部分,而语言模型则有潜力跨越这些限制,去到自然没走过的区域。
生成的序列靠谱吗?
光会“写”还不够,必须证明写出来的序列真的像是活的病毒基因组,而不是随机 DNA。
于是研究团队做了系统验证(Figure 1C–J):
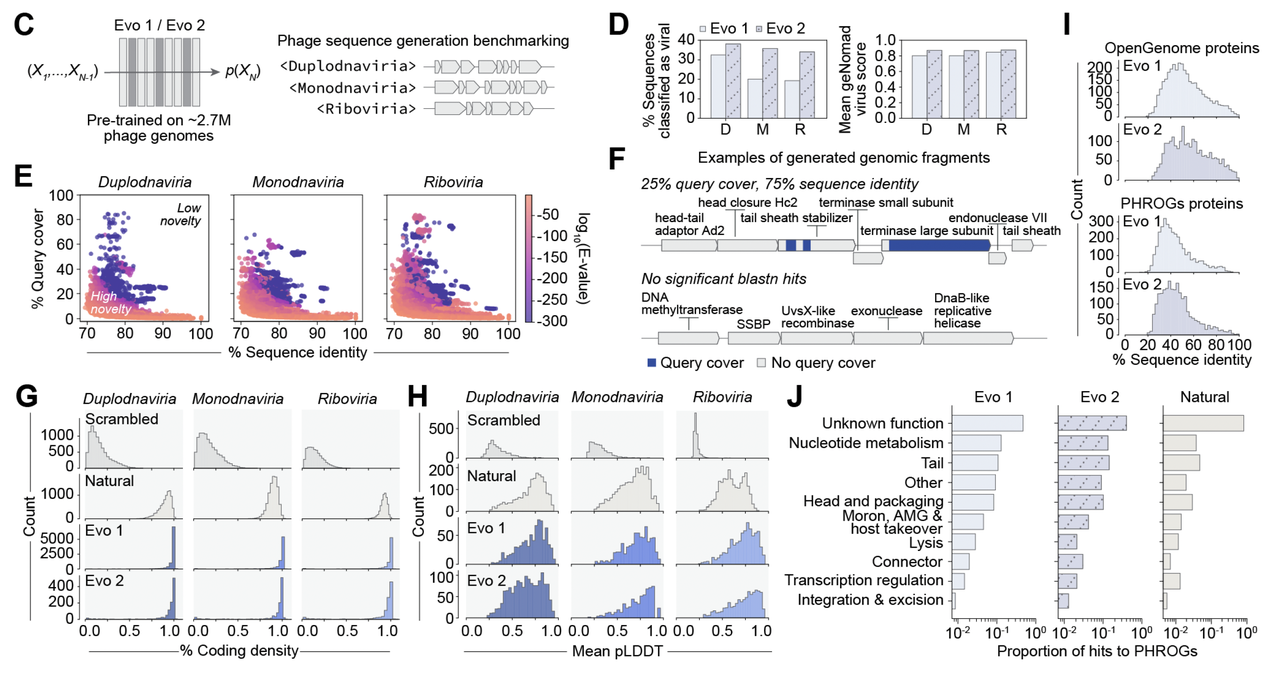
- 病毒身份确认
用 geNomad 工具分类,结果显示生成的序列大部分能被识别为病毒(Figure 1D)。
说明模型写出来的东西“像病毒”,而不是乱序列。
新颖性验证
和已知序列比对,发现相似度低,很多片段完全没有自然界对应(Figure 1E–F)。
换句话说,生成的序列不是简单抄袭,而是真正的新组合。
- 基因组特征
生成序列的编码密度(coding density)接近天然噬菌体,而远高于打乱的对照序列(Figure 1G)。
蛋白结构预测的置信度也与天然蛋白相当(Figure 1H),说明序列不仅是“字面正确”,还具备合理的折叠潜力。
- 蛋白质层面的合理性与多样性
生成蛋白和数据库里的噬菌体蛋白比对,相似性普遍很低(Figure 1I),说明有进化新颖性。
但功能注释分布却和天然噬菌体很相似(Figure 1J),意味着“功能框架”被保留了。
小结
Figure 1 整体传达的信息很明确:
语言模型真的学会了“病毒语法”,能写出结构合理的基因组;
这些基因组不是抄袭,而是有足够的新颖性;
同时在功能上和天然噬菌体保持一致。
AI 既能“写出像自然的东西”,又能“写出自然没写过的东西”。这就是 Figure 1 想展示的核心亮点。
在 Figure 1B 中,作者用图示展示了这种思路:自然进化只是探索了广阔基因组空间中的一小部分,而语言模型则有潜力跳出自然的路径,去到“未曾被探索过”的区域。
在 Figure 1C 中,研究团队通过不同提示(prompt),让模型尝试生成来自不同病毒谱系的完整序列,结果发现生成的序列能被分类工具识别为“病毒样”,而不是一堆无意义的随机 DNA。
也就是说,Evo 模型不只是复读自然的结果,而是真的学会了进化的潜在规则,因此能够创造出自然界未出现过、但依然合理的基因组。
三、如何落地?模板选择与设计流程
有了能“写基因组”的语言模型,还需要解决一个问题:怎么让它写的东西既能活,又能按照我们设定的方向去活?
设计工作流
在 Figure 2A 中,作者给出了完整的工作流:
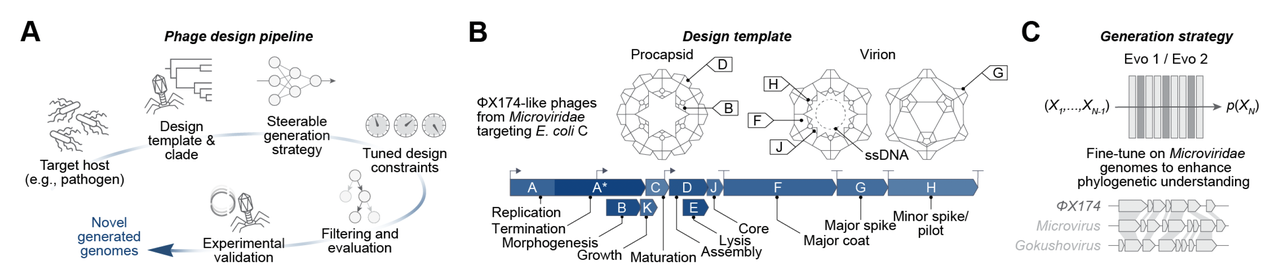
先确定一个目标宿主(比如要感染哪种细菌);
再选择一个模板噬菌体(作为“参考范本”);
用相关的基因组数据对模型进行微调;
设定设计约束(例如基因组长度范围、GC 含量、关键基因必须存在等);
让模型生成海量候选序列;
通过计算过滤掉“不合格”的,再进入实验验证环节。
这套流程的核心思想是:先给模型一个轨道,再让它自由发挥。这样既能保持“像”,又能引入“新”。
模板选择:ΦX174
在 Figure 2B 中,作者选择了一个非常经典的模板:ΦX174 噬菌体。
这是第一个被完整测序的 DNA 病毒基因组(1977 年)。
基因组很小,只有 5.4 kb,却包含 11 个基因和多个调控元件,复杂度刚好适合测试全基因组设计。
它感染的宿主是大肠杆菌 C 株,安全且研究历史丰富。
可以说,ΦX174 就像“基因组设计的Hello World”。
模型微调:专攻 Microviridae
在 Figure 2C 中,研究团队展示了如何通过 监督微调(SFT) 把 Evo 模型“定制化”,专门训练在 Microviridae 家族的序列上。这样,模型不仅会写病毒,还会写“更像 ΦX174 的病毒”。
他们发现:
如果不给提示(prompt),模型很难直接写出 ΦX174 风格的基因组;
但只要在开头给几个 ΦX174 的保守序列片段作为提示,微调后的模型就能迅速生成完整的 ΦX174 类基因组(Figure 2D–E)。
这就像写作文时,先给标题和开头一句,后面整篇文章自然就顺下来了。
设计约束:三重过滤
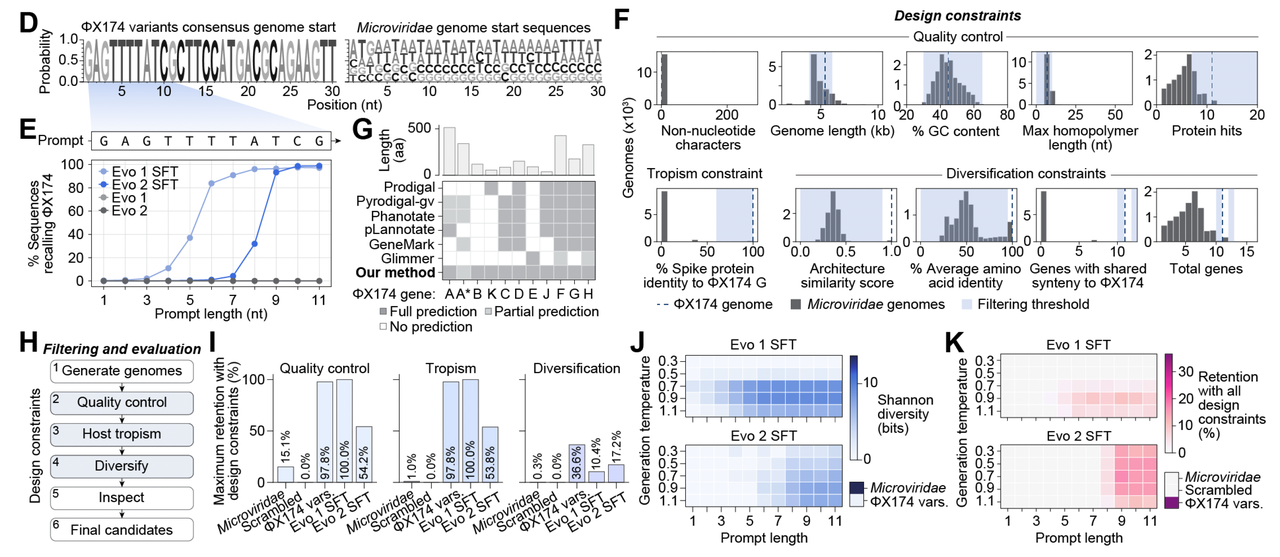
在 Figure 2F,作者提出了三类约束条件:
基本质量:长度 4–6 kb、GC 含量合适、不能有长的同碱基重复。
宿主特异性:确保生成的基因组里有和 ΦX174 足够相似的刺突蛋白(关键决定宿主范围)。
进化新颖性:要求生成的基因组和已知序列不太相似(<95% 蛋白质序列身份),同时基因排列既要保持基本框架,又允许增减一两个基因。
在 Figure 2G,团队还特别指出:现有的基因预测工具都无法完整标注 ΦX174 的 11 个基因,因为存在重叠 ORF。于是他们开发了一个专门的预测方法,确保生成的序列至少能命中大部分核心基因。
最终效果
在 Figure 2H–K,作者展示了逐层过滤的结果:
先通过基本质量控制,
再通过宿主特异性过滤,
最后应用多样性筛选。
即便经过严格的过滤,依然能保留相当比例的生成序列,而且多样性指标(香农多样性)依旧很高。
这套流程确保了:生成的基因组既像 ΦX174,又有足够的新颖性,同时还能保证功能相关性。
Figure 2 告诉我们,AI 的生成能力需要被“驯化”——先通过模板和微调,让它写在正确的轨道上;再通过层层过滤,确保生成的基因组既能活,又能进化出新东西。
四、实验验证:AI 生成的基因组真的能活
再漂亮的序列,如果只存在于计算机里,都不算“真功夫”。Figure 3 的重点就是:把 AI 生成的基因组真正合成出来,然后看它们能不能变成活生生的噬菌体。
候选基因组
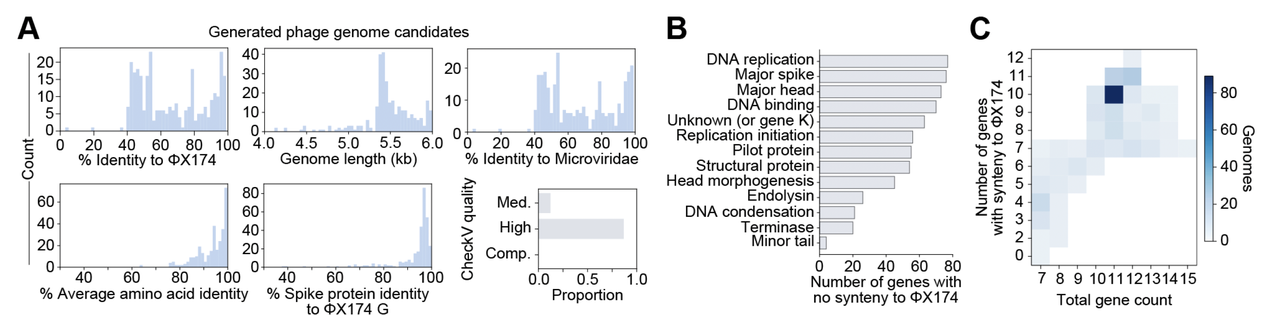
在前一轮计算筛选后,作者从模型生成的几百个序列里,挑选了 302 个候选基因组 进入实验环节(Figure 3A–C)。
这些序列的特点是:
长度 4–6 kb,和 ΦX174 相当;
平均蛋白相似度最低只有 63%,说明进化新颖性很高;
但关键的刺突蛋白仍然保持 >85% 的相似度,确保感染目标宿主的可能性。
在 Figure 3B–C 的基因组结构对比图里,我们能看到:这些序列虽然整体基因排列与 ΦX174 接近,但很多基因都有明显的“断裂”或“替换”,既保证了框架,又带来了变化。
“重启”病毒
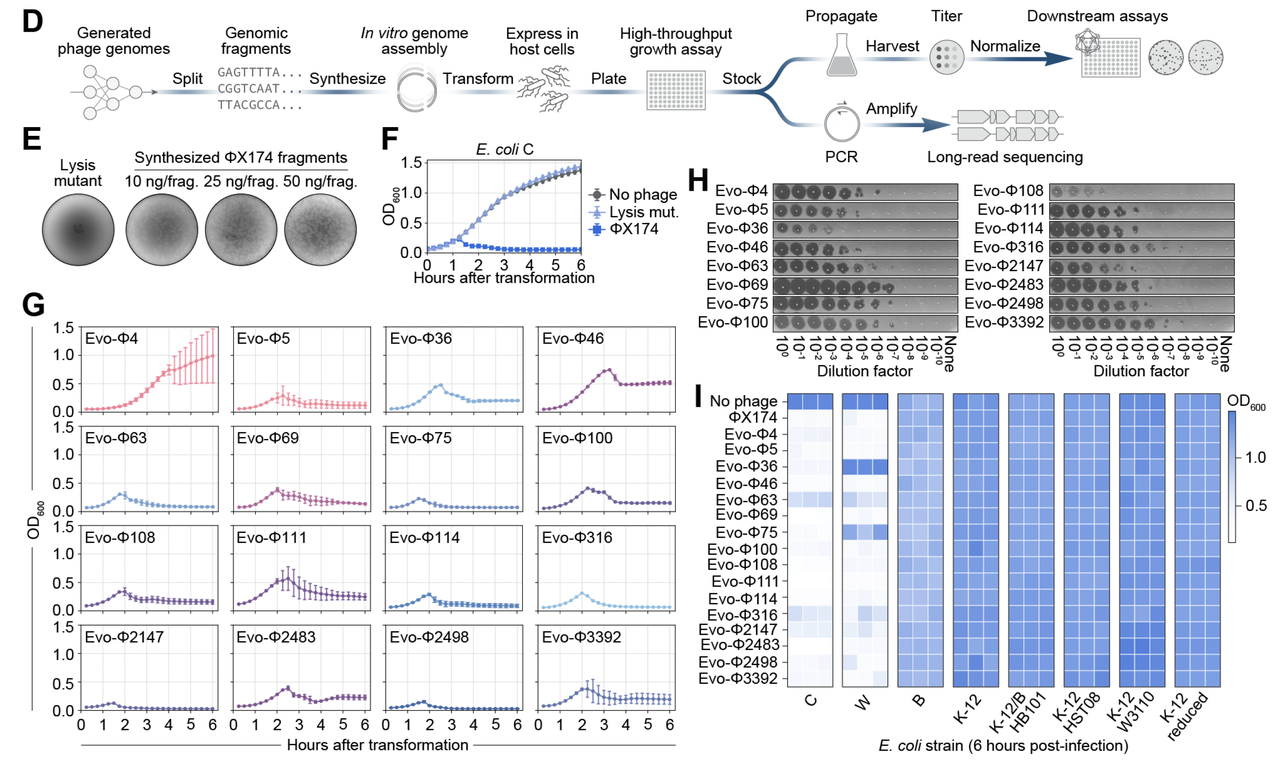
接下来是最精彩的一步:把这些序列真正合成出来,尝试“重启”成活的噬菌体。
在 Figure 3D,作者画出了完整的实验流程:先通过 DNA 合成拼接出双链 DNA,再把它转入大肠杆菌 ,让细菌的细胞机制把它包装成病毒颗粒。
这就像把一份“操作系统源码”装进硬件,看看能不能启动。
结果非常惊喜:
在 Figure 3E 的斑块实验里,可以看到这些人工基因组真的能形成一个个清晰的噬菌斑,证明病毒活了。
如果在关键裂解基因里加上失活突变,则不会形成斑块,验证了实验的可靠性。
功能测试
进一步,作者还用多种方式验证这些 AI 病毒的功能:
在 Figure 3F–G 的生长曲线上,感染过的细菌群体在短时间内被迅速抑制,和野生型 ΦX174 的效果类似。
在 Figure 3H,这些 AI 病毒能被大量扩增和滴定,说明具备完整的生命周期。
在 Figure 3I,作者检测了宿主范围:结果显示,这些病毒能够感染大肠杆菌 C(目标宿主),还能意外感染到 E. coli W,但不会感染其他 6 株非目标大肠杆菌。说明设计时设定的宿主特异性过滤是可靠的。
最终,302 个候选里有 16 个 AI 生成的基因组成功“复活”为可存活的噬菌体。
小结
Figure 3 想传达的核心信息是:
AI 写出来的并不是一堆虚拟序列,而是真的能合成出活病毒;
这些病毒不仅能感染目标宿主,还严格保持了设计时的特异性;
从计算到实验,AI 生成的基因组第一次跨越了“虚拟—现实”的鸿沟。
五、进化与结构新颖性
在拿到 16 个成功复活的 AI 噬菌体之后,研究团队并没有就此止步。他们进一步问:这些病毒的基因组和结构,到底有多新颖? Figure 4 就是答案。
基因组层面的新颖性
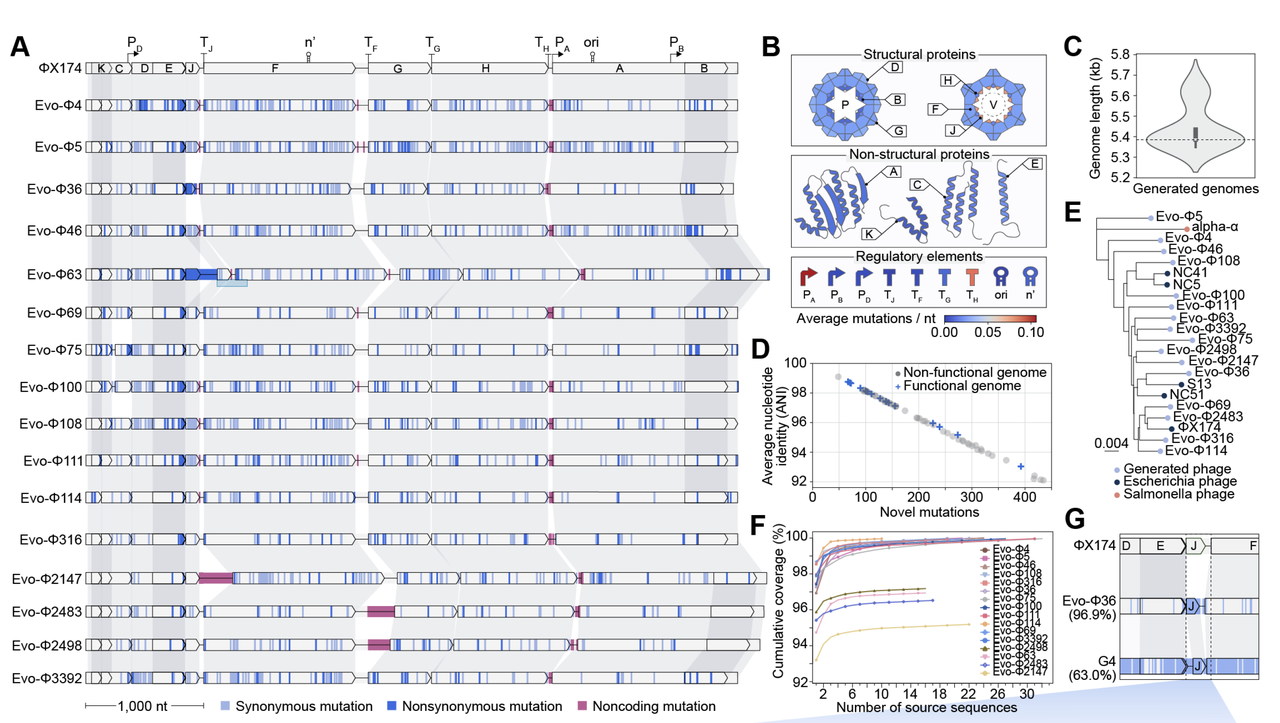
在 Figure 4A 的基因组比对图里,可以看到这些 AI 病毒和 ΦX174 的差异:上百处同义突变、非同义突变以及非编码区的变化。
其中一些变化非常显著:
Evo-Φ63 出现了一个新的 J 基因插入;
Evo-Φ114 丢失了 K 基因;
Evo-Φ75 和 Evo-Φ100 的 C 基因出现大规模缺失;
Evo-Φ4 和 Evo-Φ46 的 E 基因出现大幅延长。
这些变化在自然的进化过程中很少能稳定存在。
在 Figure 4B,作者统计了突变热点:结构蛋白 J 和一些调控元件是最容易发生变化的,而复制起点和部分终止子则完全保守。说明模型在创新的同时,仍然保留了核心“不可动”的模块。
在 Figure 4D–E 的系统发育分析里,16 个 AI 病毒和已知的 Microviridae 噬菌体形成了独立分支,有些序列和最近的自然噬菌体相比只有 93% 的核苷酸相似度,已经接近“新物种”的界定标准。
自然无法实现的组合
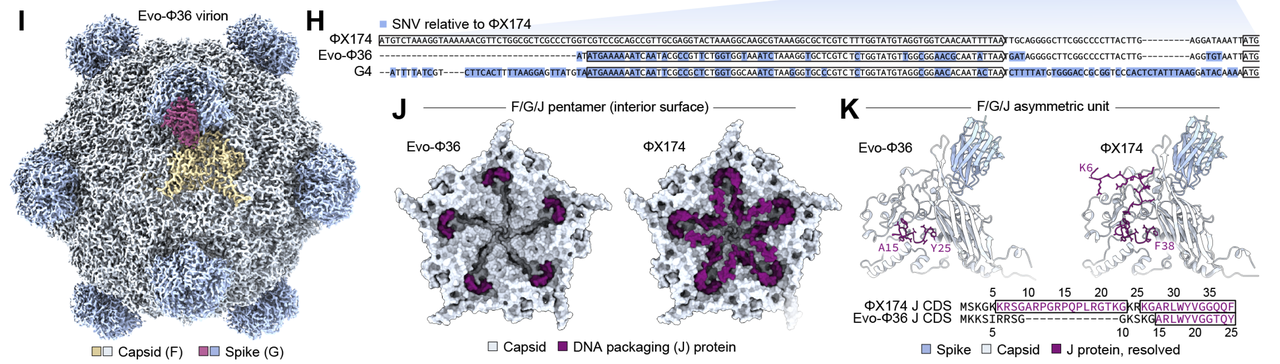
最令人兴奋的例子是 Evo-Φ36:
在 Figure 4G–H 的比对中,它的 J 基因(负责基因组装入和结构支撑)完全被来自另一个远缘噬菌体 G4 的版本替代。
按照过去的实验,如果直接把 G4 的 J 基因换到 ΦX174 里,是不可存活的。
但 AI 模型却给出了一个“上下文兼容”的组合:虽然用的是 G4 的 J 基因,但它周围的非编码区和 ΦX174 保持一致,从而让整个系统能正常工作。
结构层面的惊喜
为了验证这一点,作者用冷冻电镜(cryo-EM)解析了 Evo-Φ36 的病毒颗粒结构(Figure 4I–K):
结果发现,它的 J 蛋白和衣壳的结合方式与 ΦX174 不同,但依然保持了整体装配稳定性。
特别是 J 蛋白的 N 端不再和衣壳结合,而是更可能承担 DNA 结合功能。
这意味着模型不仅创造出了“自然界没成功过”的基因替换,还同时找到了全新的蛋白质-蛋白质协同解法。
小结
Figure 4 的核心信息是:
AI 病毒不是简单“改几个碱基”,而是真正带来了数百处基因组创新;
它们有的接近自然新物种的标准,有的甚至创造出了自然界无法稳定存在的组合;
在结构层面,AI 设计出的解法能够突破传统进化的“禁区”。
换句话说,这一步展示了 AI 生成的基因组不只是“活得下去”,而是“活得与众不同”。
六、适应性与竞争优势
活下去是一回事,活得更好又是另一回事。作者在 Figure 5 中设计了多种竞争实验,测试这些 AI 病毒在与野生型 ΦX174 的对抗中,是否具备优势。
混战实验:谁能胜出?
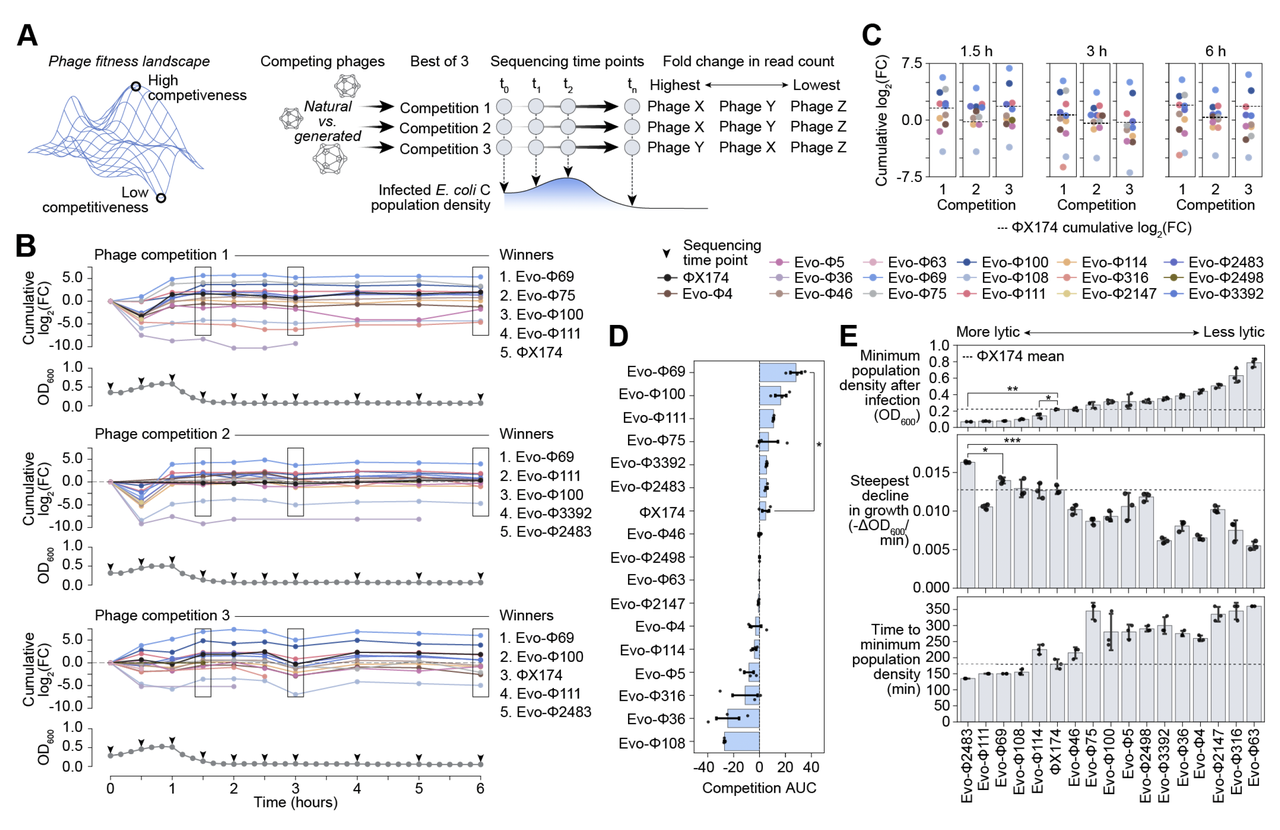
在 Figure 5A 的实验设计图中,研究团队把 16 个 AI 病毒和野生型 ΦX174 以相同剂量(MOI)混合感染同一个大肠杆菌群体。
然后通过测序追踪每个病毒种群的数量变化,累计“涨幅”最大的,就代表适应性最强。
结果很惊人(Figure 5B–D):
在三轮独立的竞争实验里,有 3 个 AI 病毒(Evo-Φ69、Evo-Φ100、Evo-Φ111) consistently 跻身前五,而 ΦX174 只在其中两轮进入前五,最高也只能排第三。
其中 Evo-Φ69 表现最亮眼,在所有实验里都稳居第一,感染 6 小时后种群数量增加了 16–65 倍,而 ΦX174 的涨幅只有 1.3–4 倍。
这说明 AI 不仅能“复制”一个能活的病毒,还能创造出比自然原型更具竞争力的新病毒。
裂解动力学:谁杀菌更快?
除了混战,作者还单独测量了每个病毒的“杀伤效率”(Figure 5E):
通过跟踪感染后的细菌生长曲线,他们比较了不同病毒的裂解速度和强度。
其中 Evo-Φ2483 裂解效率最快最强,能在 135 分钟内把细菌群体降到极低水平,而 ΦX174 需要 180 分钟才能做到,且最低细胞密度还更高。
其他几个 AI 病毒(Evo-Φ111、Evo-Φ69、Evo-Φ108)也都表现出比 ΦX174 更强的裂解效果。
关键发现
有的 AI 病毒在混战中胜出(适应性高);
有的 AI 病毒在裂解速度上领先(杀伤力强);
不同 AI 病毒展现了多样化的“性能优势”,说明它们不只是简单模仿,而是探索出了不同的进化路径。
小结
Figure 5 的核心信息是:
AI 病毒的适应性和杀菌效率,在多个维度上超过了天然 ΦX174;
这表明生成模型不仅能复刻自然,还能高效探索自然未必能轻易到达的“高性能区域”。
换句话说,AI 在病毒设计上实现了“功能超越”,创造出了一批比自然更强的候选者。
七、AI 病毒如何克服耐药性
噬菌体最大的应用潜力之一,就是用来对抗耐药细菌。但问题在于:细菌进化出耐药性往往很快,而天然噬菌体往往“一招鲜”,遇到耐药菌株就完全失效。
Figure 6 展示了一个非常关键的发现:AI 病毒在克服耐药性方面展现出天然噬菌体做不到的优势。
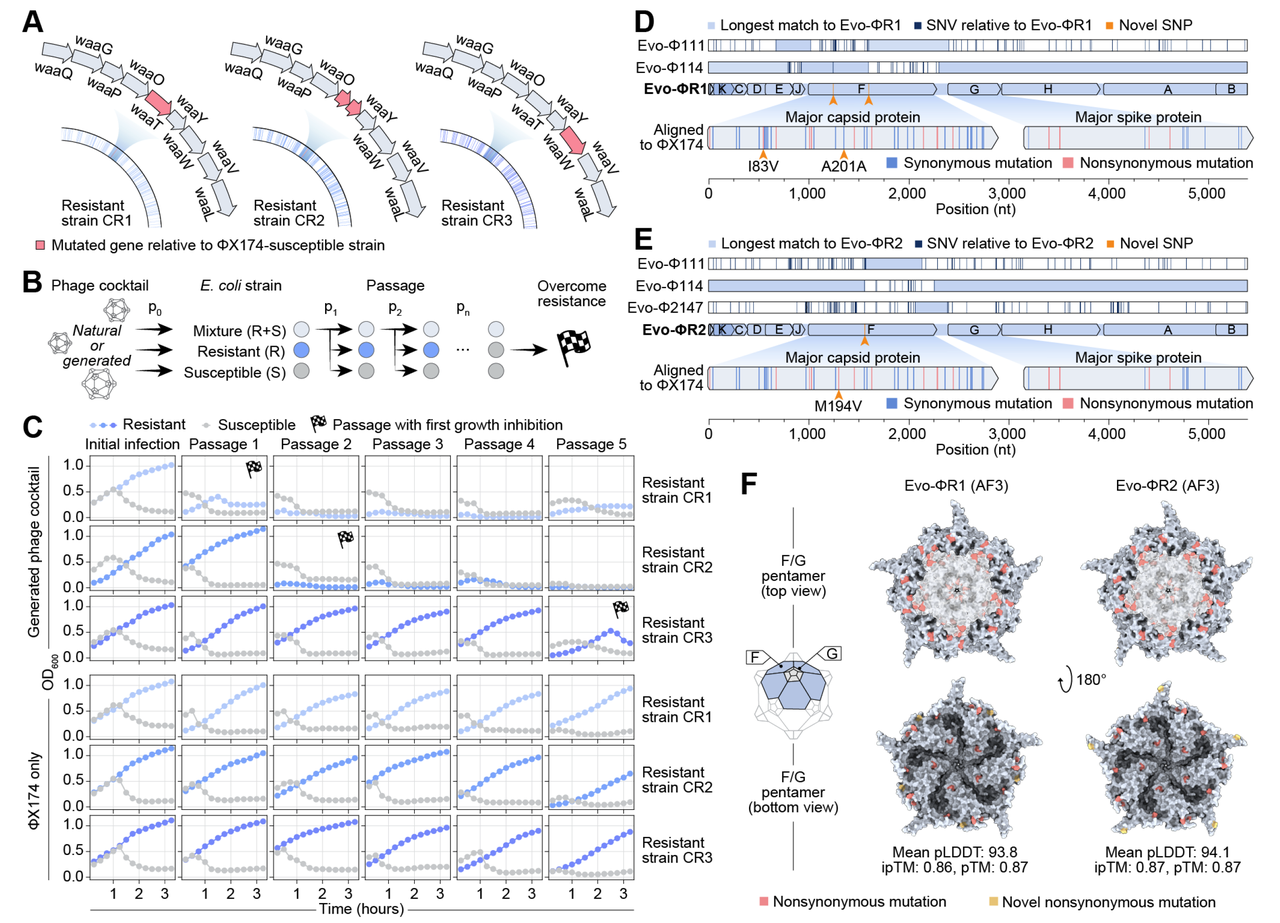
耐药菌株的挑战
作者首先构建了三种对野生型 ΦX174 耐药的大肠杆菌 C 突变株(Figure 6A–B)。
实验证明:这些菌株对 ΦX174 完全没有反应,ΦX174 感染后几乎不产生任何噬菌斑(Figure 6C)。
换句话说,天然 ΦX174 在这些耐药菌面前彻底“报废”。
AI 病毒的突破
然后,研究团队测试了 16 个 AI 病毒在这些耐药菌株上的表现(Figure 6D–E):
单个 AI 病毒往往在某些耐药菌株上依旧失败,但不同 AI 病毒的感染谱并不重叠。
有的能感染耐药株 1,但不能感染耐药株 2;有的则反之。
于是,作者尝试了一个“病毒鸡尾酒”的思路(Figure 6F):
把 16 个 AI 病毒混合在一起,用来同时攻击三株耐药细菌。
结果显示:混合后的 AI 病毒组合,几乎完全消除了所有耐药菌群体。
对比之下,野生型 ΦX174 即使加量也完全无效。
核心意义
这说明:
AI 病毒的多样性 本身就是一种优势 —— 它们覆盖了更广的宿主谱。
当它们组合在一起,就能像“精准打击小分队”一样,全面应对不同的耐药突变。
这为未来设计 可定制的噬菌体鸡尾酒疗法 提供了新思路,有潜力应用于对抗抗生素耐药危机。
小结
Figure 6 的信息很直接:
天然 ΦX174 遇到耐药菌完全无能为力;
但 AI 生成的噬菌体凭借多样化的基因组创新,能够 集体突破耐药屏障。
这意味着,AI 病毒不仅是“新奇的科学展示”,更可能成为未来抗菌治疗中的 实用武器。
八、总结与展望
从 Figure 1 到 Figure 6,这篇文章完整展示了一条令人振奋的路径:
从语言模型的“虚拟生成”(学会进化语法,写出像样的基因组),
到实验室的“真实验证”(302 个候选 → 16 个成功复活),
再到功能和应用的“性能超越”(更强的适应性、杀菌效率,以及克服耐药性)。
这是第一次,人类真正依靠 AI 生成并“重启”了全新的噬菌体基因组。
这项工作的意义远不止于病毒本身:
它证明了 AI 可以从蛋白质设计跨越到全基因组设计,生命系统的复杂性不再是不可逾越的障碍。
它为 个性化抗菌疗法 打开了可能性:未来,我们或许可以针对不同的耐药细菌,用 AI 即时生成噬菌体“鸡尾酒”。
它更是宣告了一个新方向:生成式基因组学(generative genomics)。
在文章的讨论部分,作者用了很克制的语言。但从学界的眼光看,这是一个真正意义上的范式转变:
过去我们只能微调自然已有的基因组;
现在,AI 已经可以帮助我们探索自然未曾走过的“生命空间”。
这不仅是一次技术突破,更是一次哲学式的跃迁:
我们第一次看到,AI 可以作为“进化的代理人”,与自然共同探索生命的可能性。
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →