是妥协,也是突破|RFdiffusion3:RFdiffusion进入全原子设计时代
在蛋白质设计领域,扩散模型的引入堪称一次革命。2023 年,Baker 团队在 Nature 发表的 RFdiffusion(RFD1) 为 de novo 蛋白设计提供了一条全新的路径:只要给出功能性约束,就能在计算机中“长出”全新的蛋白质结构。利用rosetta系列工具做出的一系列设计,也让David Baker获得了2024年的诺贝尔奖。随后推出的 RFdiffusion2(RFD2) 又进一步解决了部分活性位点嵌入的问题,让一些简单的催化功能得以实现。
不过,前两代方法仍然存在明显的瓶颈:
精度不足:它们主要在残基层面建模,无法直接处理每一个原子的位置,这让小分子结合、DNA 识别等原子级任务依然难以实现。
限制较多:设计时只能对少数关键原子进行约束,无法全面控制氢键、溶剂可及性或复杂的非蛋白组分。

就在昨天(2025年9月18日),华盛顿大学 David Baker 团队在 bioRxiv 上发表了最新工作 《De novo Design of All-atom Biomolecular Interactions with RFdiffusion3》。在这篇文章中,研究人员提出了 RFdiffusion3(RFD3) ——一个全原子级的扩散模型,不仅能生成蛋白主链和侧链,还能同时处理小分子、DNA 等非蛋白分子。相比前代,它在计算效率上提升了一个数量级,同时在蛋白–蛋白、蛋白–DNA、蛋白–小分子结合以及酶设计等多类任务中全面超越了旧有方法。
这项工作标志着蛋白质设计进入了一个新阶段:从残基到原子,设计的分辨率第一次与分子相互作用的真实尺度对齐。
🔗原文链接:https://doi.org/10.1101/2025.09.18.676967
一、方法与架构:全原子扩散的实现
RFdiffusion3 的最大创新,是把扩散建模的基本单元从“残基”提升到“原子”。这意味着,模型不仅关心主链如何折叠,还同时追踪每一个侧链原子的空间坐标。在此基础上,它还能在同一个体系中处理非蛋白质分子,例如小分子、DNA 或 RNA。
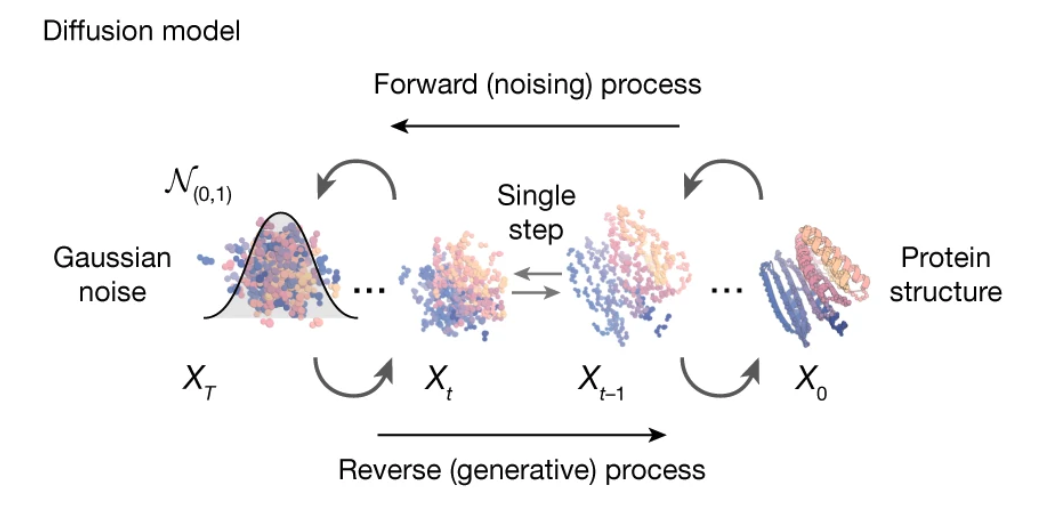
图 1a:扩散过程

这一部分展示了 RFD3 如何从完全噪声出发,逐步还原出有序的蛋白质结构。不同时间点的快照让人直观感受到:原子级扩散并不是“一下子生成”,而是一个不断消除噪声、逐渐逼近真实结构的过程。与前两代相比,这次不仅骨架在收敛,侧链的细节也在同步生成。
图 1b:适用范围

这张图像是一份功能清单:RFD3 能覆盖蛋白–蛋白相互作用、蛋白–DNA 结合、蛋白–小分子结合、酶活性位点设计以及对称性寡聚体生成。过去这些任务往往需要不同工具分别处理,而现在通过一个统一的架构就能实现,体现了模型的通用性。
图 1c:模型架构
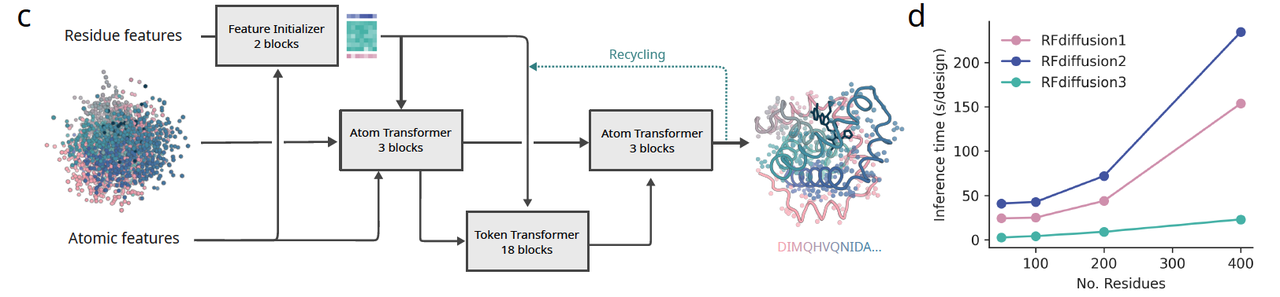
RFD3 采用了一个轻量化的 transformer–U-Net 架构:
特征下采样 → 把原子和残基的局部信息编码;
稀疏注意力 → 强调几何邻近的原子之间的交互;
跨层注意力 → 在原子级和残基层特征之间建立联系;
特征上采样 → 输出每个原子的空间坐标更新。
图 1d:速度对比
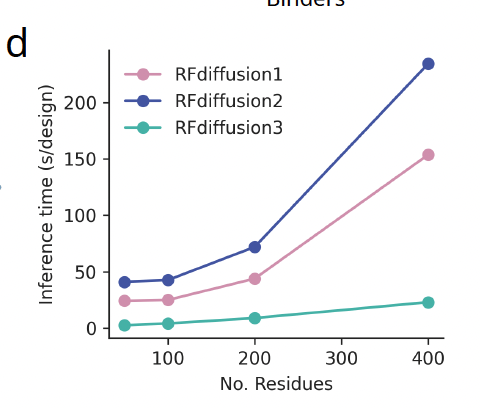
速度是另一个重要亮点。RFD3 在相同任务下比 RFD2 快了约 10 倍,这一点对实际应用意义重大。它意味着研究人员可以在更短时间里生成和筛选更多设计,提高整个设计–实验循环的效率。
整体来看,Figure 1 向读者传达了一个核心信息:RFD3 不是局部改良,而是一次从架构到任务范围的全面升级。
三、条件控制与可编程性:让设计进入“原子操作”时代
如果说全原子扩散让 RFD3 拥有了分辨率,那么条件控制则赋予了它“可编程”的能力。研究人员可以在生成过程中直接设定原子层面的几何或化学条件,从而引导模型生成满足特定功能的结构。
图 2a:酶活性位点的扩散
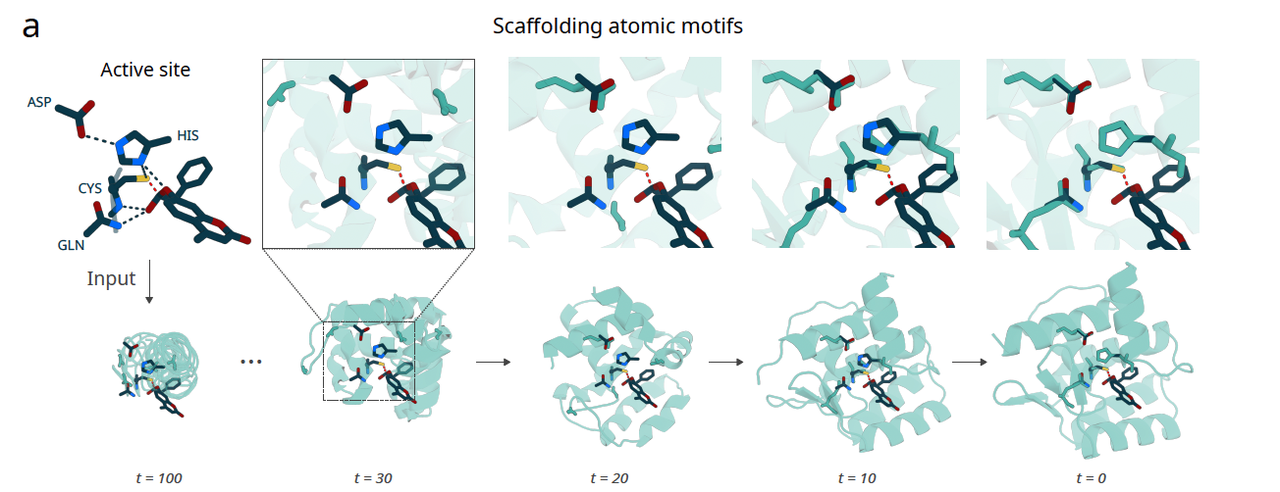
这一部分展示了 RFD3 如何处理酶催化所需的原子集合。模型不是简单地把活性位点固定在某个残基上,而是让网络在扩散过程中“自己决定”最合适的残基来承担这些原子。结果是:活性位点自然嵌入蛋白骨架,形成合理的化学环境。
图 2b–c:蛋白–DNA与蛋白–小分子共同生成
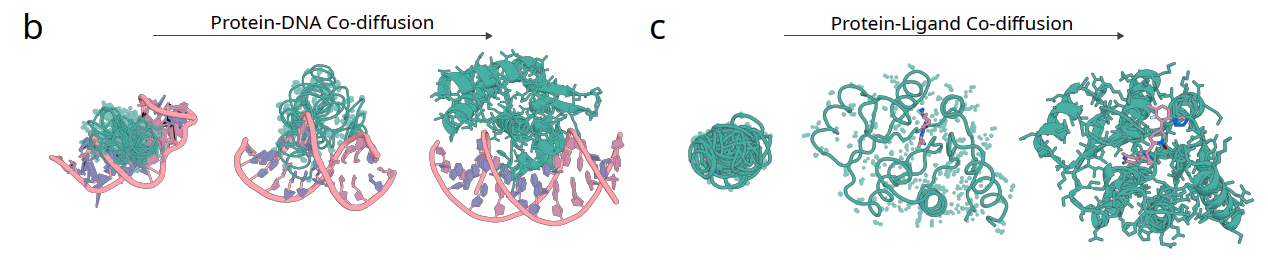
传统方法往往把 DNA 或小分子看作固定的外部对象,只设计蛋白部分。而在 RFD3 中,DNA 构象或小分子构象可以和蛋白一起被扩散生成。这样,结合界面不再是硬对接,而是动态适配,最终得到更加自然的相互作用模式。
图 2d:氢键约束
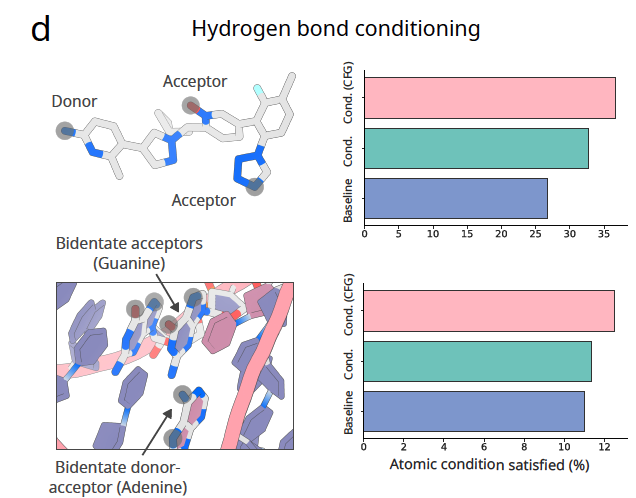
设计蛋白–小分子或蛋白–DNA 结合时,研究者可以明确指定哪些原子要作为氢键供体或受体。图中结果显示,有了这种约束后,氢键形成的比例显著提高。
图 2e:溶剂可及性控制
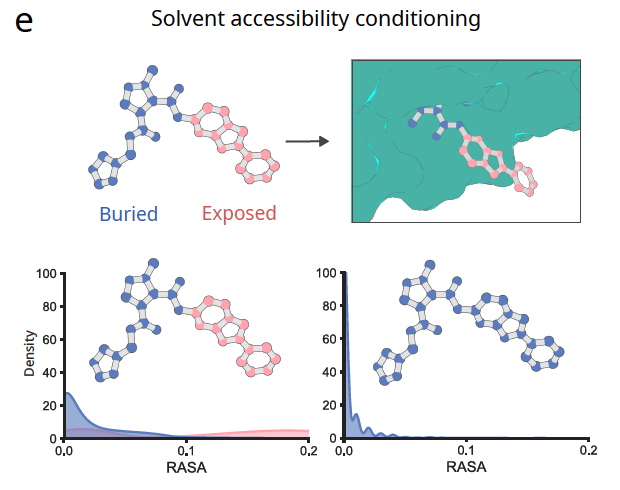
通过给出原子的溶剂可及性(RASA),模型可以决定某个小分子是否深埋在蛋白内部,或者暴露在表面。这为调节结合口袋的“开放”程度提供了直接手段。
图 2f:质心位置
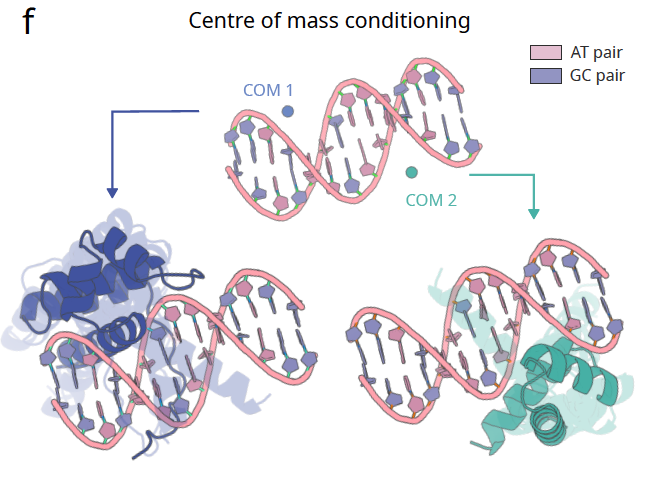
设计者甚至可以控制蛋白质整体相对于配体的空间位置。例如在 DNA 结合任务中,可以设定不同的蛋白质质心,模型会自动调整生成的构象来匹配。
图 2g:对称性约束
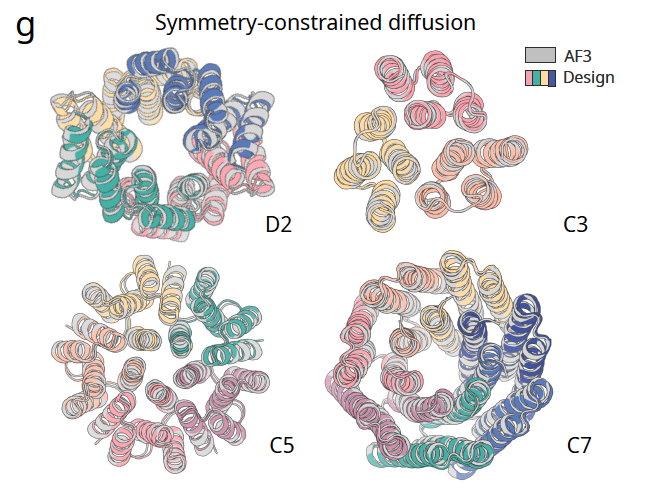
只要在扩散输入中加入对称噪声,RFD3 就能生成符合指定对称群的蛋白寡聚体。图中展示了 D2、C3、C5、C7 等对称性设计,结果与 AlphaFold3 的预测高度一致。
Figure 2 展现的,是 RFD3 从“会生成”到“能被精确操控”的关键跨越。它让蛋白设计真正具备了像编程一样的自由度:研究者不再只是被动等待模型输出,而是能在原子层面对结构施加精细指令。
四、设计任务上的表现:全面超越前代
有了全原子扩散和灵活的条件控制,RFD3 在一系列核心任务上的表现都大幅提升。Figure 3 用四组对比展示了它的优势。
图 3a:蛋白–蛋白结合
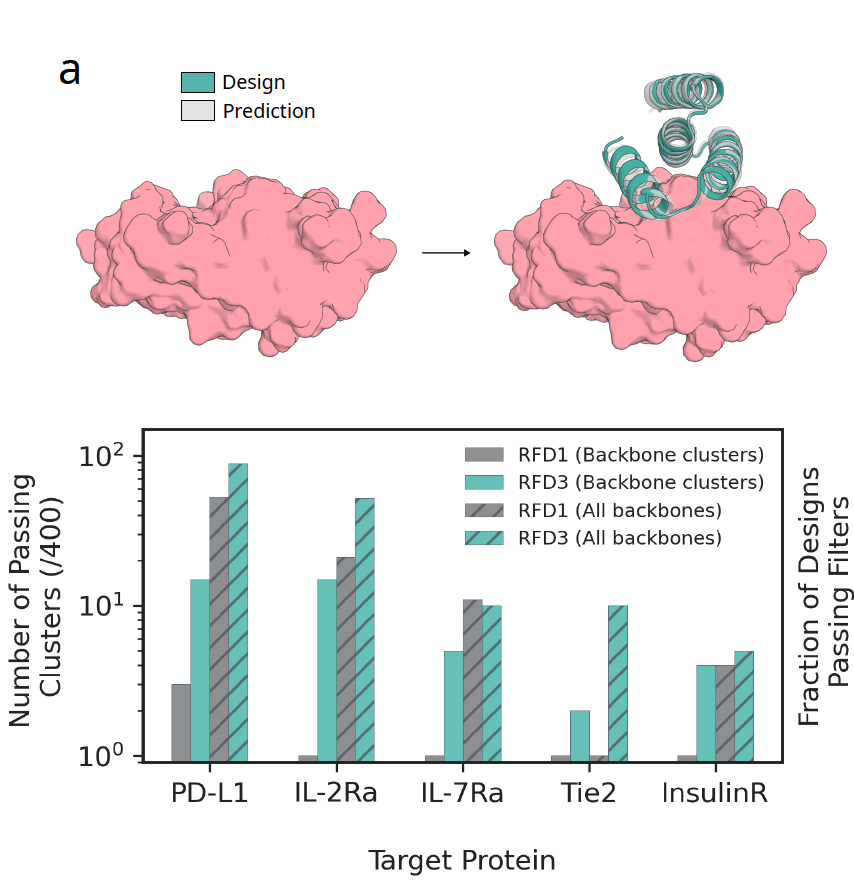
研究团队把 RFD3 和 RFD1 在五个经典结合靶标上进行比较(PD-L1、IL-2Ra、IL-7Ra、Tie2、InsulinR)。结果显示,RFD3 不仅成功率更高,而且能找到更多结构多样化的解决方案。平均每个靶标能得到 8.2 个成功簇,而 RFD1 只有 1.4 个。这意味着,RFD3 不再局限于单一构象,而是能探索出多条可能的结合路径。
图 3b:蛋白–DNA结合
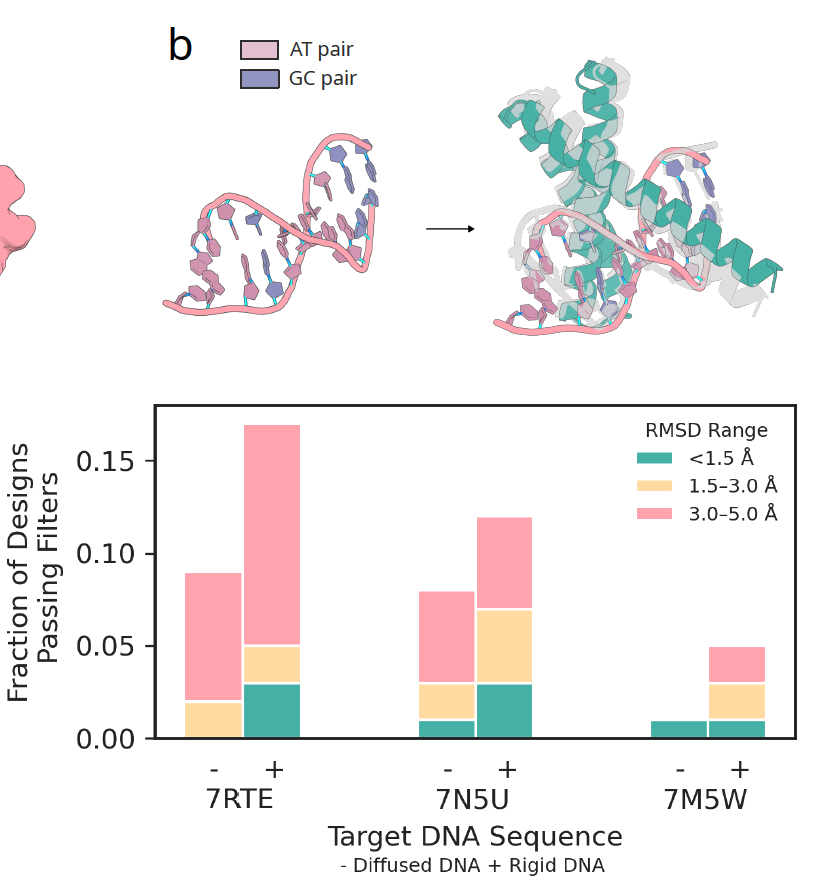
DNA 结合一直是设计难点,因为 DNA 构象本身灵活、极性强。RFD3 的做法是同时生成 DNA 构象和蛋白界面。测试结果表明,即便不给定 DNA 的结构(只给出序列),RFD3 依然能生成合理的结合界面,并保持在 5 Å RMSD 以内的精度。这是此前方法几乎无法做到的。
图 3c:小分子结合
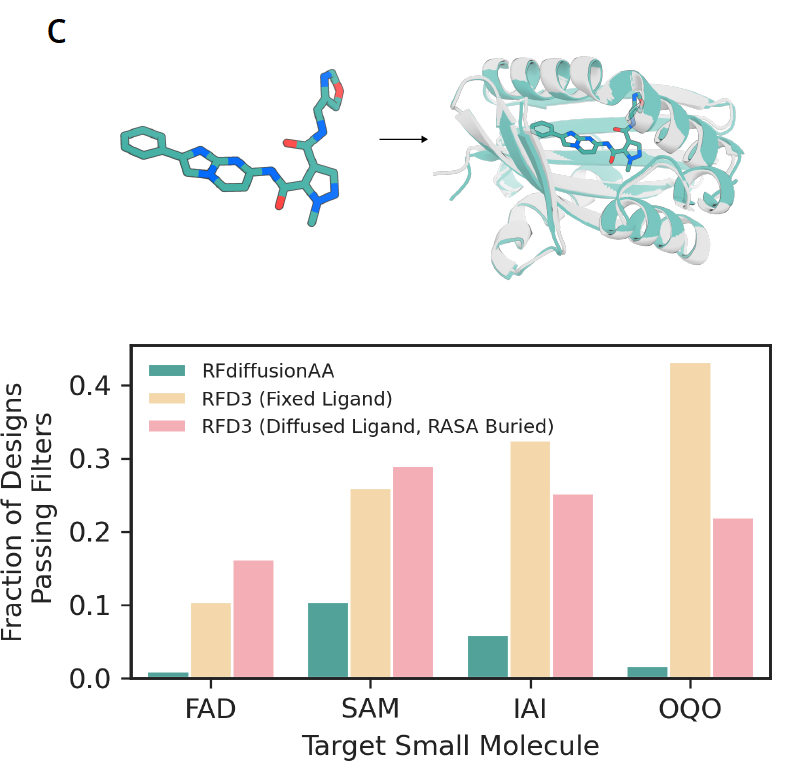
过去的扩散模型往往需要固定小分子的构象再来设计蛋白。RFD3 则能把小分子与蛋白一起扩散生成,捕捉到不同的结合姿态。对四个小分子(包括常见的 FAD、SAM,以及不常见的 IAI、OQO)的测试中,RFD3 全面优于上一代方法,不仅成功率高,还能给出更低的结合能量和更丰富的新颖结构。
图 3d:酶设计

酶设计需要对活性位点的原子精确定位。在 Atomic Motif Enzyme 基准测试中,RFD3 在 41 个活性位点任务里,有 90% 的案例超过了 RFD2。尤其是在包含多个残基岛(分散的残基片段)的复杂情境中,RFD3 的成功率远高于前代。这说明它能够更好地在复杂骨架中嵌入活性中心。
Figure 3 的四个子图合在一起,传递的核心信息非常直接:无论是结合蛋白、核酸、小分子,还是催化活性位点,RFD3 都在成功率、结构多样性和能量学合理性上全面胜出。
五、实验验证:从计算机到现实
再漂亮的计算机模型,如果不能在实验室里验证,就只能停留在理论。研究团队专门挑选了两个最能体现 RFD3 原子级优势的挑战:DNA 结合蛋白设计和酶设计,并把结果带到了实验台上。
图 4a–b:DNA 结合蛋白
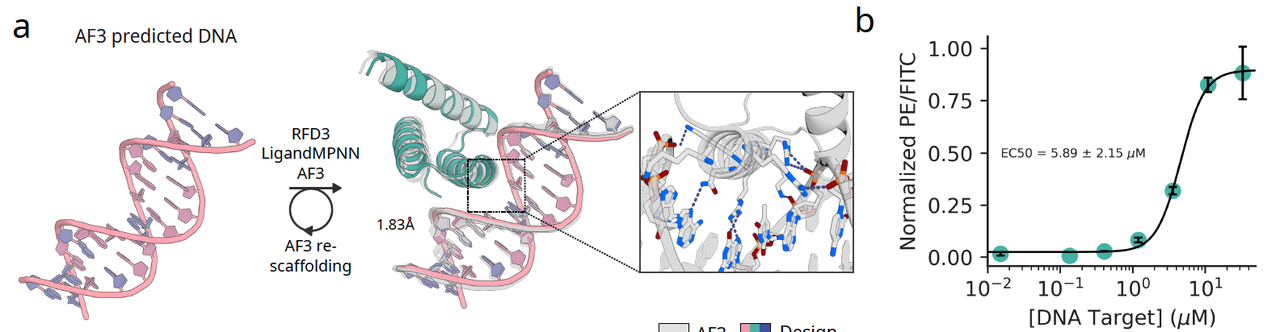
研究者首先用 RFD3 对一段随机生成的 DNA 序列进行设计。在第一轮生成后,他们选择了预测可靠的构象,再通过“重支架化”方法优化结合区,最终得到 5 个候选设计。将这些设计表达并检测结合活性后,结果发现其中有一个能稳定结合目标 DNA,且达到 低微摩尔级亲和力(EC50 ~ 5.9 μM)。
在蛋白质设计领域,这是一个相当有说服力的验证:模型不仅能设计出能“折叠”的结构,还能精确识别特定 DNA 序列。
图 4c–d:酶设计
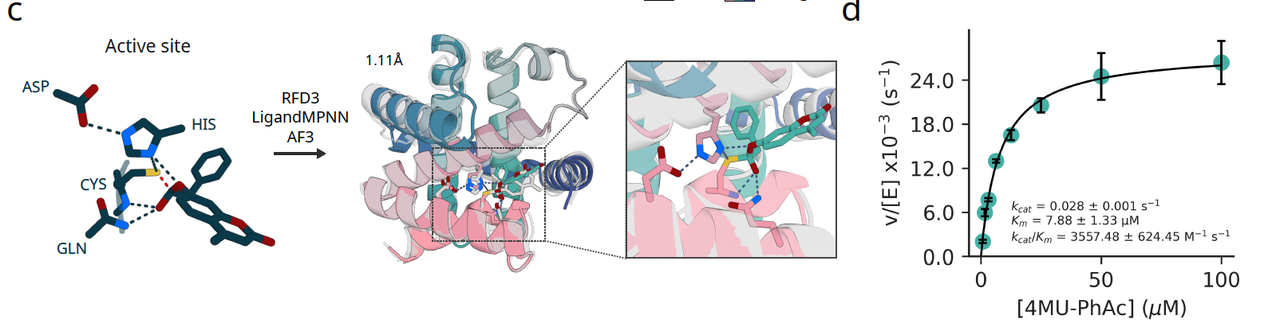
第二个测试是一个更具挑战性的任务:设计一类半胱氨酸水解酶,它需要经典的 Cys–His–Asp 催化三联体来完成底物水解。研究团队用 RFD3 根据最小的活性原子集合进行 scaffolding,随后在实验中检测设计的催化活性。
在 190 个设计中,有 35 个表现出多轮催化活性,其中最优的一个酶达到 Kcat/Km = 3557,超过了之前使用 RFD2 的成果。
这意味着 RFD3 在催化中心的原子排布上足够精确,能够直接产生活性。
Figure 4 展示的,不只是两个实验结果,而是一个完整的闭环:
从计算机上的原子级扩散;
到模型生成合理的结合界面或活性位点;
再到实验室验证其功能确实可行。
这一闭环让 RFD3 的价值更加清晰:它不只是一个漂亮的算法,而是一个可以真正加速分子设计周期的实用工具。
六、总结与展望:原子级的未来
RFdiffusion3 的出现,让蛋白质设计第一次真正进入了原子级的时代。它具备三个核心特征:
分辨率:以原子为建模单元,不再停留在残基层面;
可编程性:研究者可以直接设定氢键、溶剂可及性、对称性等条件,引导设计走向目标;
效率:在全面提升精度的同时,比前一代方法快了一个数量级。
从 Figure 1 到 Figure 4,我们看到的不仅是模型架构的升级,更是任务范围和实验结果的全面突破:
蛋白–蛋白结合更加多样;
蛋白–DNA 结合首次实现同时生成 DNA 构象;
小分子结合不再局限于固定构象;
酶设计成功率显著提高;
实验验证证明它能真正产生活性分子。
这些进展让我们能够想象一个全新的场景:未来在药物研发、合成生物学、工业酶设计中,研究者或许可以像编程一样,直接在原子层面“写下”自己想要的功能,然后由模型生成候选分子,再快速走向实验室测试。
但同时也可以看到的是,所谓的All-atom设计,其实也并不够“All-atom",蛋白质的翻译后修饰、糖蛋白的设计等任务都没有涉及。而且,David Baker也是做了妥协,RFD3采用了Alphafold3来代替Rosettafold-AllAtom,所以可能性能的提升是来自于结构预测工具的改变,而在这一点上,又有一点为别人做嫁衣的感觉。
不过,作为用户,我们希望的还是能用上一个更好的设计工具,能够针对具体的科学问题,得到我们想要的结果,在这一点上,看起来RFD3还是非常具有突破性的!
延伸阅读
本文属于 AI4S文献 栏目。
返回 AI4S文献 → 去公众号阅读完整版 →